Introduction
L’interférence ARN (ARNi) est un mécanisme de régulation de la plupart des cellules eucaryotes qui utilise de petites molécules d’ARN double brin (ARNdb) comme déclencheurs pour contrôler directement l’activité des gènes en fonction de leur homologie (Figure 1) (1). Connues sous le nom de petits ARN interférents (siRNA), ces molécules d’ARNd ∼21-22 pb de long ont des surplombs 3′ de 2 nt caractéristiques qui leur permettent d’être reconnues par la machinerie enzymatique de l’ARNi, ce qui conduit finalement à la dégradation homologique-dépendante de l’ARNm cible. Dans les cellules de mammifères, les siRNA sont produits à partir du clivage de précurseurs d’ARNdb plus longs par l’endonucléase RNase III Dicer (2), ou ils peuvent être synthétisés par des méthodes chimiques ou biochimiques. Dicer est complexé avec des protéines de liaison à l’ARN, la protéine de liaison à l’ARN TAR (TRBP), PACT et Ago-2, qui sont impliquées dans le transfert des siRNA au complexe de silençage induit par l’ARN (RISC) (3). Les composants essentiels du RISC sont les membres de la famille Argonaute (Ago). Chez l’homme, il existe huit membres de cette famille, mais seul Ago-2 possède un domaine catalytique actif pour l’activité de clivage (4,5). Alors que les siRNA chargés dans le RISC sont à double brin, Ago-2 clive et libère le brin » passager « , ce qui conduit à une forme activée du RISC avec une molécule d’ARN » guide » à simple brin qui dirige la spécificité de la reconnaissance de la cible par appariement intermoléculaire des bases (6). Les règles qui régissent la sélectivité du chargement du brin dans le RISC sont basées sur les stabilités thermodynamiques différentielles des extrémités des siRNA (7,8). L’extrémité la moins stable thermodynamiquement est favorisée pour la liaison au domaine PIWI de Ago-2.
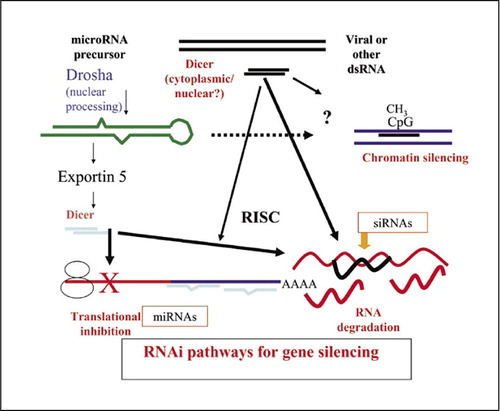
L’interférence ARN est multiforme, et il existe diverses voies dans lesquelles de petits ARN double brin (ARNdb) régulent l’expression des gènes. La voie des micro-ARN endogènes (miRNA) commence par des miRNA primaires transcrits par Pol II qui sont transformés dans le noyau en pré-miRNA, exportés vers le cytoplasme et transformés à nouveau en miRNA fonctionnels. La fonction principale des miARN est d’inhiber la traduction via un appariement de base Watson-Crick incomplet aux régions 3′ non traduites des ARNm ciblés. Par ailleurs, de petits ARN interférents (siRNA) parfaitement duplexés peuvent être produits de manière intracellulaire ou fournis de manière exogène aux cellules. Le brin guide est incorporé dans le complexe de silençage induit par l’ARN (RISC), où il guide la dégradation spécifique de la séquence du transcrit cible, indépendamment de l’endroit où se produit l’appariement des bases. Les voies du miRNA et du siRNA sont interchangeables, et les déterminants importants sont les positions dans le message et l’étendue de l’appariement des bases avec les transcrits ciblés. Les siRNA peuvent également déclencher l’extinction transcriptionnelle des gènes via des interactions avec la chromatine, où ils guident la méthylation des histones et de l’ADN conduisant à une chromatine inactive.
MicroARN
Un bras important de l’ARNi implique les microARN (miRNA). Il s’agit de duplex endogènes qui régulent de manière posttranscriptionnelle l’expression des gènes en se complexant avec RISC et en se liant aux régions 3′ non traduites (UTR) des séquences cibles via de courts tronçons d’homologie, appelés » séquences graines » (9,10). Le principal mécanisme d’action des miRNA est la répression traductionnelle, bien que celle-ci puisse s’accompagner d’une dégradation du message (11). Les duplex de miARN possèdent un appariement de bases Watson-Crick incomplet, et le brin antisens ne peut pas être choisi par clivage du brin passager comme c’est le cas pour les siARN ; le brin antisens doit donc être choisi par un mécanisme alternatif (12-14). Les miARN sont des substrats endogènes pour la machinerie ARNi. Ils sont initialement exprimés sous forme de longs transcrits primaires (pri-miRNAs), qui sont transformés dans le noyau en épingles à cheveux de 60-70 pb par le complexe Microprocesseur, composé de Drosha et DGCR8 (15,16) en pré-miRNAs. Les pré-miARN sont ensuite traités dans le cytoplasme par Dicer et l’un des deux brins est chargé dans le RISC, vraisemblablement via une interaction avec l’une des protéines accessoires de Dicer (3). Il est important de noter qu’il est possible d’exploiter cette voie d’extinction génétique native pour réguler le ou les gènes de son choix. Si l’effecteur siRNA est délivré à la cellule, il » activera » le RISC, ce qui entraînera une extinction puissante et spécifique de l’ARNm ciblé. En raison de la puissance et de la sélectivité de l’ARNi, il est devenu la méthodologie de choix pour réduire au silence l’expression de gènes spécifiques dans les cellules de mammifères.
L’ARNi comme approche thérapeutique pour le traitement des maladies
Le contrôle des gènes associés aux maladies fait de l’ARNi un choix attrayant pour les thérapeutiques futures. Pratiquement toutes les maladies humaines causées par l’activité d’un ou de quelques gènes devraient pouvoir être traitées par l’ARNi. Cette liste comprend le cancer, les maladies auto-immunes, les troubles génétiques dominants et les infections virales. L’ARNi peut être déclenché par deux voies différentes : (i) une approche basée sur l’ARN, dans laquelle les siRNA effecteurs synthétiques sont délivrés par divers transporteurs aux cellules cibles sous forme de duplex de 21 bases préformés ; ou (ii) des stratégies basées sur l’ADN, dans lesquelles les siRNA effecteurs sont produits par traitement intracellulaire de transcrits ARN en épingle à cheveux plus longs (voir références 17 et 18). Cette dernière approche repose principalement sur la synthèse nucléaire d’ARN à épingle à cheveux courte (shRNA), qui sont transportés vers le cytoplasme par la voie d’exportation des miRNA et sont transformés en siRNA par Dicer. Si l’utilisation directe d’effecteurs synthétiques d’ARNsi est simple et donne généralement lieu à une puissante extinction des gènes, l’effet est transitoire. Les médicaments ARNi basés sur l’ADN, en revanche, ont le potentiel d’être introduits de manière stable lorsqu’ils sont utilisés dans un contexte de thérapie génique, ce qui permet, en principe, un traitement unique de gènes shRNA délivrés par un vecteur viral.
Les premières applications cliniques de l’ARNi ont été dirigées vers le traitement de la dégénérescence maculaire liée à l’âge (DMLA), qui provoque la cécité ou une vision limitée chez des millions d’adultes chaque année (19,20). Des thérapies basées sur l’ARNi sont également en cours de développement pour les infections virales, notamment le virus de l’immunodéficience humaine (VIH), les virus de l’hépatite B et C (VHB et VHC) et le virus respiratoire syncytial (VRS) (21). Des stratégies pour le traitement des maladies neurodégénératives et des cancers sont également en bonne voie.
Bien que des études in vivo réussies aient montré l’efficacité potentielle des thérapies basées sur l’ARNi, d’autres études ont illustré des approches spécifiques à éviter lors de l’adoption d’un mécanisme cellulaire endogène pour un bénéfice thérapeutique. Parmi les effets secondaires indésirables, citons l’activation des récepteurs de type Toll (TLR) et des réponses à l’interféron de type 1, ainsi que la concurrence avec les composants endogènes de la voie ARNi (22). Ces résultats indiquent que, bien que l’ARNi soit potentiellement un mécanisme révolutionnaire pour le traitement des maladies, il faut faire preuve de la prudence nécessaire lors de l’interprétation des résultats des knockdowns de cibles médiés par l’ARNi.
Le défi de l’administration de siRNA spécifiques aux cellules ou aux tissus est également crucial lors de l’étude de l’utilité des thérapies basées sur l’ARNi pour une maladie donnée ; diverses stratégies d’administration non virale et virale de déclencheurs ARNi se sont révélées efficaces dans leurs modèles de maladies respectifs. Les avantages et inconvénients relatifs de l’utilisation de siRNA synthétiques par rapport aux shRNA exprimés doivent également être pris en considération lors de la conception de thérapies basées sur l’ARNi pour une maladie particulière.
Les siRNA synthétisés chimiquement sont couramment criblés pour un knockdown efficace d’un gène cible spécifique. Pour augmenter la stabilité des siRNA, des modifications chimiques sont introduites, telles que des 2′-O-méthylpurines ou des 2′-fluoropyrimidines (23). Lors de la conception initiale d’une molécule de siRNA, on utilise couramment des algorithmes de calcul qui intègrent divers paramètres, notamment les stabilités des extrémités du duplex de siRNA pour une sélection correcte des brins et les structures secondaires de l’ARNm pour l’accessibilité au site cible. Pour améliorer la puissance d’une réponse ARNi, les duplex d’ARNsi peuvent également être conçus pour imiter des substrats pour le traitement par Dicer (24). Les siRNA plus longs (par exemple, 27-mères) sont incorporés dans l’étape de chargement de Dicer de la voie ARNi et peuvent faciliter l’activation de RISC. Les 27-mères sont conçus de manière asymétrique pour présenter un surplomb 3′ de 2 nt à une extrémité et une région émoussée à l’autre (25), ce qui guide le traitement par Dicer et la biogenèse du brin guide approprié, puisque le domaine PAZ de Dicer reconnaît l’extrémité du surplomb. En outre, en raison de l’efficacité des 27-mères dans la médiation de l’extinction des gènes, une concentration plus faible de siRNA peut médier une réponse ARNi puissante.
Du point de vue des applications thérapeutiques de l’ARNi, la préoccupation la plus importante est la livraison des siRNA au tissu approprié. De nombreuses publications récentes ont montré que les siRNA peuvent être délivrés de manière systémique à divers tissus avec un knockdown résultant des ARN cibles. L’injection intraveineuse de siRNA en vue d’une administration systémique est réalisée par la conjugaison de molécules de siRNA à un groupe cholestérol ou par l’emballage de siRNA dans des particules liposomales. L’administration systémique utilisant ces approches est efficace pour l’administration au foie et au jéjunum, mais peut ne pas être appropriée pour l’administration à d’autres organes. Dans une étude de preuve de concept, des siRNA ciblant l’apolipoprotéine B (APOB) ont été utilisés pour modifier le métabolisme du cholestérol. Le groupe 3′ hydroxyle sur le brin passager du siRNA a été chimiquement lié à un groupe cholestérol, et ces siRNA conjugués ont effectivement fait chuter l’expression du gène de >50 % dans le foie et de 70 % dans le jéjunum (26).
Une autre approche pour l’administration systémique implique l’utilisation de bicouches lipidiques spécialisées appelées particules stables acide nucléique-lipide (SNALP), qui incorporent des siRNA chimiquement modifiés (27). Des lipides cationiques et neutres composent la bicouche, ainsi qu’un revêtement hydrophile externe de polyéthylène glycol (PEG). Dans une étude, des singes ont reçu une dose unique de SNALP contenant des siRNA, ce qui a permis de réduire le taux de cholestérol pendant 11 jours ou plus, avec <10% de l’expression de l’APOB restant dans le foie de ce modèle de primate non humain (28). Aucune toxicité notable n’a été observée, ce qui suggère l’utilité potentielle de cette méthode dans la délivrance systémique.
Pour l’efficacité in vivo des molécules de siRNA, le dosage des siRNA délivrés est une considération pratique, et la délivrance sélective de siRNA à des tissus spécifiques permettrait potentiellement de réduire le dosage efficace requis. Le ciblage des récepteurs de la surface cellulaire est une approche avantageuse, car elle permet de réduire la dose de siRNA et d’éviter potentiellement les effets hors cible de la délivrance de siRNA à des tissus non pertinents. Le couplage des siRNA à des aptamères ou à des fragments d’anticorps, ou l’utilisation de nanoparticules recouvertes de ligands spécifiques aux récepteurs, permet de délivrer spécifiquement les charges utiles de siRNA aux cellules et tissus ciblés. Pour cibler les cellules infectées par le VIH, les siRNA ont été couplés à des fragments d’anticorps à chaîne lourde (Fab) qui reconnaissent la glycoprotéine d’enveloppe du VIH gp120. De la protamine chargée positivement a été conjuguée aux molécules Fab, et les siRNA chargés négativement ont interagi électrostatiquement avec la protamine pour former un complexe Fab-siRNA. Cette approche basée sur les anticorps a démontré >70% de knockdown de la protéine antigène spécifique du groupe p24 (Gag) lors du ciblage de lymphocytes T cultivés infectés par le VIH-1 (29). Une autre approche de ciblage a tiré parti d’un peptide du virus de la rage, qui se lie spécifiquement au récepteur de l’acétylcholine. Lorsque ce peptide a été conjugué à un peptide polyarginine qui se lie aux siRNA, la livraison des siRNA au système nerveux central a été accomplie, entraînant l’inhibition d’une infection virale encéphalitique mortelle (30).
Les aptamères, qui sont des ligands ARN structurés, peuvent être conçus pour se lier spécifiquement aux récepteurs de la surface cellulaire et être liés de manière covalente aux siRNA pour une livraison in vivo spécifique. Une méthode a utilisé des aptamères qui se lient à l’antigène membranaire spécifique de la prostate (PSMA) exprimé à la surface des cellules cancéreuses de la prostate. Conjugués à des siRNA, ces hybrides aptamère-siRNA ont réduit efficacement la croissance tumorale chez la souris (31). Une approche similaire utilisant à la fois des siRNA biotinylés et des aptamères liés à la streptavidine, une protéine liant la biotine, a fait appel à des siRNA de 27-mer pour induire puissamment l’extinction des gènes (32).
L’enrobage de nanoparticules avec des ligands spécifiques du type de cellule est une autre approche puissante pour délivrer systématiquement des molécules induisant des ARNi. Dans une importante étude de preuve de concept, les tumeurs du sarcome d’Ewing ont été ciblées in vivo avec des nanoparticules enrobées de ligand de transferrine (33). Ces nanoparticules ont été construites à l’aide de polycations contenant de la cyclodextrine (CDP) spécifiquement conçus pour incorporer des molécules de siRNA chargées négativement. Pour plus de stabilité et pour prévenir l’agrégation, des polymères PEG ont été attachés à la surface extérieure en utilisant des groupes terminaux adamantane. Les ligands de transferrine ont ensuite été liés de manière covalente aux chaînes adamantane-PEG, et la conception des nanoparticules a permis leur auto-assemblage en nanoparticules uniformes de ∼50 nanomètres. Les siRNA incorporés dans les nanoparticules ciblaient le produit de fusion génétique Ews-Fli1 (Ewing sarcoma breakpoint region 1-flightless 1 homolog) et il a été démontré qu’ils inhibaient la formation de tumeurs l chez la souris (33).
Marques de conclusion
En résumé, la progression de la découverte initiale de l’ARNi à ses applications cliniques a été stupéfiante. La compréhension de la biologie fondamentale de l’ARNi a conduit à ses applications généralisées dans la recherche fondamentale, puis dans les applications pour le traitement des maladies. Au cours des prochaines années, nous devrions nous attendre à découvrir davantage de régulation de l’expression des gènes par l’ARNi et à voir des médicaments à base d’ARNi approuvés pour le traitement de maladies. En outre, l’ARNi s’est révélé être un outil puissant pour l’étude de la fonction des gènes et a ouvert de nouveaux domaines de recherche fondamentale. Dans un avenir proche, nous devrions assister à un développement continu de notre compréhension et de l’application de ce mécanisme cellulaire remarquable de régulation posttranscriptionnelle de l’expression des gènes.
Remerciements
Ce travail a été soutenu par l’Institut national des allergies et des maladies infectieuses des Instituts nationaux de la santé et l’Institut du cœur, des poumons et du sang des NIH à J.J.R. D.H.K. est soutenu par une bourse de pré-doctorat City of Hope.
Déclaration d’intérêts concurrents
Les auteurs ne déclarent aucun intérêt concurrent.
- 1. Almeida, R. et R.C. Allshire. 2005. L’ARN silencieux et la régulation du génome. Trends Cell Biol. 15:251-258.Crossref, Medline, CAS, Google Scholar
- 2. Zhang, H., F.A. Kolb, L. Jaskiewicz, E. Westhof, et W. Filipowicz. 2004. Single processing center models for human Dicer and bacterial RNase III. Cell 118:57-68.Crossref, Medline, CAS, Google Scholar
- 3. Lee, Y., I. Hur, S.Y. Park, Y.K. Kim, M.R. Suh, et V.N. Kim. 2006. The role of PACT in the RNA silencing pathway. EMBO J. 25:522-532.Crossref, Medline, CAS, Google Scholar
- 4. Meister, G., M. Landthaler, A. Patkaniowska, Y. Dorsett, G. Teng, et T. Tuschl. 2004. Human Argonaute2 mediates RNA cleavage targeted by miRNAs and siRNAs. Mol. Cell 15:185-197.Crossref, Medline, CAS, Google Scholar
- 5. Liu, J., M.A. Carmell, F.V. Rivas, C.G. Marsden, J.M. Thomson, J.J. Song, S.M. Hammond, L. Joshua-Tor, et al. 2004. Argonaute2 est le moteur catalytique de l’ARNi des mammifères. Science 305:1437-41.Crossref, Medline, CAS, Google Scholar
- 6. Tang, G. 2005. siRNA and miRNA : an insight into RISCs. Trends Biochem. Sci. 30:106-114.Crossref, Medline, CAS, Google Scholar
- 7. Schwarz, D.S., G. Hutvagner, T. Du, Z. Xu, N. Aronin, et P.D. Zamore. 2003. Asymmetry in the assembly of the RNAi enzyme complex. Cell 115:199-208.Crossref, Medline, CAS, Google Scholar
- 8. Khvorova, A., A. Reynolds, et S.D. Jayasena. 2003. Functional siRNAs and miRNAs exhibit strand bias. Cell 115:209-216.Crossref, Medline, CAS, Google Scholar
- 9. Bartel, D.P. 2004. MicroRNAs : genomics, biogenesis, mechanism, and function. Cell 116:281-297.Crossref, Medline, CAS, Google Scholar
- 10. Bartel, D.P. et C.Z. Chen. 2004. Micromanagers de l’expression des gènes : l’influence potentiellement étendue des microARN métazoaires. Nat. Rev. Genet. 5:396-400.Crossref, Medline, CAS, Google Scholar
- 11. Bagga, S., J. Bracht, S. Hunter, K. Massirer, J. Holtz, R. Eachus, et A.E. Pasquinelli. 2005. Regulation by let-7 and lin-4 miRNAs results in target mRNA degradation. Cell 122:553-563.Crossref, Medline, CAS, Google Scholar
- 12. Leuschner, P.J., S.L. Ameres, S. Kueng, et J. Martinez. 2006. Clivage du brin passager du siRNA pendant l’assemblage du RISC dans les cellules humaines. EMBO Rep. 7:314-320.Crossref, Medline, CAS, Google Scholar
- 13. Gregory, R.I., T.P. Chendrimada, N. Cooch, et R. Shiekhattar. 2005. Human RISC couples microRNA biogenesis and posttranscriptional gene silencing. Cell 123:631-640.Crossref, Medline, CAS, Google Scholar
- 14. Matranga, C., Y. Tomari, C. Shin, D.P. Bartel, et P.D. Zamore. 2005. Passenger-strand cleavage facilitates assembly of siRNA into Ago2-containing RNAi enzyme complexes. Cell 123:607-620.Crossref, Medline, CAS, Google Scholar
- 15. Lee, Y., C. Ahn, J. Han, H. Choi, J. Kim, J. Yim, J. Lee, P. Provost, et al. 2003. The nuclear RNase III Drosha initiates microRNA processing. Nature 425:415-419.Crossref, Medline, CAS, Google Scholar
- 16. Han, J., Y. Lee, K.H. Yeom, Y.K. Kim, H. Jin, et V.N. Kim. 2004. The Drosha-DGCR8 complex in primary microRNA processing. Genes Dev. 18:3016-3027.Crossref, Medline, CAS, Google Scholar
- 17. Hannon, G.J. et J.J. Rossi. 2004. Débloquer le potentiel du génome humain avec l’interférence ARN. Nature 431:371-378.Crossref, Medline, CAS, Google Scholar
- 18. Scherer, L.J. et J.J. Rossi. 2003. Approches pour le knockdown spécifique à la séquence de l’ARNm. Nat. Biotechnol. 21:1457-1465.Crossref, Medline, CAS, Google Scholar
- 19. Fattal, E. et A. Bochot. 2006. Délivrance oculaire d’acides nucléiques : oligonucléotides antisens, aptamères et siRNA. Adv. Drug Deliv. Rev. 58:1203-1223.Crossref, Medline, CAS, Google Scholar
- 20. Tolentino, M. 2006. La technologie de l’ARN d’interférence dans le traitement du CNV. Ophthalmol. Clin. North Am. 19:393-399 vi-vii.Medline, Google Scholar
- 21. Leonard, J.N. et D.V. Schaffer. 2006. Thérapie ARNi antivirale : approches émergentes pour frapper une cible mobile. Gene Ther. 13:532-540.Crossref, Medline, CAS, Google Scholar
- 22. Svoboda, P. 2007. Off-targeting et autres effets non spécifiques des expériences RNAi dans les cellules de mammifères. Curr. Opin. Mol. Ther. 9:248-257.Medline, CAS, Google Scholar
- 23. Czauderna, F., M. Fechtner, S. Dames, H. Aygun, A. Klippel, G.J. Pronk, K. Giese, et J. Kaufmann. 2003. Structural variations and stabilising modifications of synthetic siRNAs in mammalian cells. Nucleic Acids Res. 31:2705-2716.Crossref, Medline, CAS, Google Scholar
- 24. Kim, D.H., M.A. Behlke, S.D. Rose, M.S. Chang, S. Choi, et J.J. Rossi. 2005. Synthetic dsRNA Dicer substrates enhance RNAi potency and efficacy. Nat. Biotechnol. 23:222-226.Crossref, Medline, CAS, Google Scholar
- 25. Amarzguioui, M., P. Lundberg, E. Cantin, J. Hagstrom, M.A. Behlke, et J.J. Rossi. 2006. Rational design and in vitro and in vivo delivery of Dicer substrate siRNA. Nat. Protocols 1:508-517.Crossref, Medline, CAS, Google Scholar
- 26. Soutschek, J., A. Akinc, B. Bramlage, K. Charisse, R. Constien, M. Donoghue, S. Elbashir, A. Geick, et al. 2004. Therapeutic silencing of an endogenous gene by systemic administration of modified siRNAs. Nature 432:173-178.Crossref, Medline, CAS, Google Scholar
- 27. Morrissey, D.V., J.A. Lockridge, L. Shaw, K. Blanchard, K. Jensen, W. Breen, K. Hartsough, L. Machemer, et al. 2005. Potent and persistent in vivo anti-HBV activity of chemically modified siRNAs. Nat. Biotechnol. 23:1002-1007.Crossref, Medline, CAS, Google Scholar
- 28. Zimmermann, T.S., A.C. Lee, A. Akinc, B. Bramlage, D. Bumcrot, M.N. Fedoruk, J. Harborth, J.A. Heyes, et al. 2006. RNAi-mediated gene silencing in non-human primates. Nature 441:111-114.Crossref, Medline, CAS, Google Scholar
- 29. Song, E., P. Zhu, S.K. Lee, D. Chowdhury, S. Kussman, D.M. Dykxhoorn, Y. Feng, D. Palliser, et al. 2005. Antibody mediated in vivo delivery of small interfering RNAs via cell-surface receptors. Nat. Biotechnol. 23:709-717.Crossref, Medline, CAS, Google Scholar
- 30. Kumar, P., H. Wu, J.L. McBride, K.E. Jung, M.H. Kim, B.L. Davidson, S.K. Lee, P. Shankar, et al. 2007. Transvascular delivery of small interfering RNA to the central nervous system. Nature 448:39-43.Crossref, Medline, CAS, Google Scholar
- 31. McNamara, J.O., II, E.R. Andrechek, Y. Wang, K.D. Viles, R.E. Rempel, E. Gilboa, B.A. Sullenger, et P.H. Giangrande. 2006. Cell type-specific delivery of siRNAs with aptamer-siRNA chimeras. Nat. Biotechnol. 24:1005-1015.Crossref, Medline, CAS, Google Scholar
- 32. Chu, T.C., K.Y. Twu, A.D. Ellington, et M. Levy. 2006. Aptamer mediated siRNA delivery. Nucleic Acids Res. 34:e73.Crossref, Medline, Google Scholar
- 33. Hu-Lieskovan, S., J.D. Heidel, D.W. Bartlett, M.E. Davis, et T.J. Triche. 2005. Sequence-specific knockdown of EWS-FLI1 by targeted, nonviral delivery of small interfering RNA inhibits tumor growth in a murine model of metastatic Ewing’s sarcoma. Cancer Res. 65:8984-8992.Crossref, Medline, CAS, Google Scholar
.