Introduzione
L’interferenza dell’RNA (RNAi) è un meccanismo di regolazione della maggior parte delle cellule eucariotiche che utilizza piccole molecole di RNA a doppio filamento (dsRNA) come trigger per il controllo diretto omologia-dipendente dell’attività genica (Figura 1) (1). Conosciuti come piccoli RNA interferenti (siRNA), queste molecole di dsRNA lunghe ∼21-22 bp hanno caratteristiche sporgenze di 2 nt in 3′ che permettono loro di essere riconosciuti dal macchinario enzimatico dell’RNAi, che alla fine porta alla degradazione omologia-dipendente dell’mRNA target. Nelle cellule dei mammiferi, i siRNA sono prodotti dalla scissione di precursori dsRNA più lunghi da parte dell’endonucleasi RNase III Dicer (2), o possono essere sintetizzati con metodi chimici o biochimici. Dicer è complessato con le proteine leganti l’RNA, la TAR-RNA-binding protein (TRBP), PACT e Ago-2, che sono coinvolte nel passaggio dei siRNA al complesso di silenziamento indotto dall’RNA (RISC) (3). I componenti principali del RISC sono i membri della famiglia degli Argonauti (Ago). Negli esseri umani ci sono otto membri di questa famiglia, ma solo Ago-2 possiede un dominio catalitico attivo per l’attività di scissione (4,5). Mentre i siRNA caricati nel RISC sono a doppio filamento, Ago-2 scinde e rilascia il filamento “passeggero”, portando ad una forma attivata di RISC con una molecola di RNA “guida” a filamento singolo che dirige la specificità del riconoscimento del bersaglio attraverso l’accoppiamento di basi intermolecolari (6). Le regole che governano la selettività del caricamento del filamento nel RISC si basano sulla stabilità termodinamica differenziale delle estremità dei siRNA (7,8). L’estremità meno stabile termodinamicamente è favorita per il legame al dominio PIWI di Ago-2.
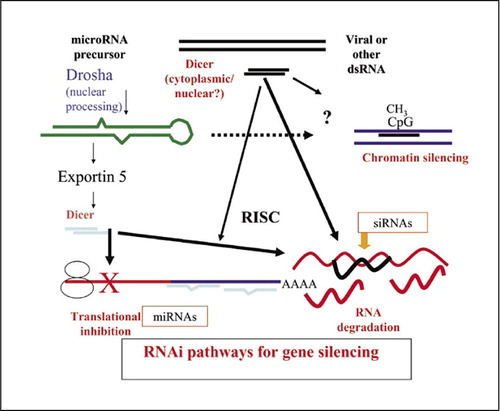
RNAi è multiforme, e ci sono varie vie in cui piccoli RNA a doppio filamento (dsRNA) regolano l’espressione genica. Il percorso endogeno dei micro-RNA (miRNA) inizia con i miRNA primari trascritti da Pol II che vengono trasformati nel nucleo in pre-miRNA, esportati nel citoplasma e trasformati nuovamente in miRNA funzionali. La funzione primaria dei miRNA è quella di inibire la traduzione attraverso l’accoppiamento di base Watson-Crick incompleto alle regioni non tradotte 3′ degli mRNA mirati. In alternativa, piccoli RNA interferenti (siRNA) perfettamente duplexati possono essere prodotti intracellularmente o forniti esogenamente alle cellule. Il filamento guida è incorporato nel complesso di silenziamento indotto dall’RNA (RISC), dove guida la degradazione sequenza-specifica del trascritto bersaglio, indipendentemente da dove si verifica l’accoppiamento di basi. Le vie del miRNA e del siRNA sono intercambiabili, e i fattori determinanti sono le posizioni all’interno del messaggio e la misura dell’accoppiamento di base con i trascritti bersaglio. I siRNA possono anche innescare il silenziamento trascrizionale dei geni attraverso le interazioni con la cromatina, dove guidano la metilazione degli istoni e del DNA portando alla cromatina inattiva.
MicroRNA
Un importante braccio di RNAi coinvolge i microRNA (miRNA). Questi sono duplex endogeni che regolano post-trascrizionalmente l’espressione genica complessando con RISC e legandosi alle regioni non tradotte 3′ (UTR) delle sequenze target attraverso brevi tratti di omologia, chiamati “sequenze seme” (9,10). Il principale meccanismo d’azione dei miRNA è la repressione traslazionale, anche se questa può essere accompagnata dalla degradazione del messaggio (11). I duplex di miRNA possiedono un accoppiamento di basi Watson-Crick incompleto, e il filamento antisenso non può essere scelto tramite scissione del filamento passeggero come avviene per i siRNA; quindi il filamento antisenso deve essere scelto da un meccanismo alternativo (12-14). I miRNA sono substrati endogeni per il macchinario RNAi. Sono inizialmente espressi come lunghi trascritti primari (pri-miRNA), che sono elaborati all’interno del nucleo in hairpin di 60-70 bp dal complesso Microprocessor, composto da Drosha e DGCR8 (15,16) in pre-miRNA. I pre-miRNA vengono ulteriormente elaborati nel citoplasma da Dicer e uno dei due filamenti viene caricato nel RISC, presumibilmente attraverso l’interazione con una delle proteine accessorie Dicer (3). È importante notare che è possibile sfruttare questo percorso di silenziamento genico nativo per la regolazione di uno o più geni a scelta. Se l’effettore siRNA viene consegnato alla cellula, esso “attiverà” RISC, con conseguente silenziamento potente e specifico dell’mRNA mirato. A causa della potenza e della selettività della RNAi, è diventata la metodologia di scelta per il silenziamento dell’espressione genica specifica nelle cellule di mammifero.
RNAi come approccio terapeutico per il trattamento delle malattie
Il controllo dei geni associati alle malattie rende la RNAi una scelta attraente per le terapie future. Fondamentalmente ogni malattia umana causata dall’attività di uno o pochi geni dovrebbe essere suscettibile di un intervento basato sull’RNAi. Questa lista include il cancro, le malattie autoimmuni, i disturbi genetici dominanti e le infezioni virali. La RNAi può essere attivata da due diversi percorsi: (i) un approccio basato sull’RNA in cui gli effettori sintetici siRNA sono consegnati da vari vettori alle cellule bersaglio come duplex di 21 basi preformati; o (ii) attraverso strategie basate sul DNA in cui gli effettori siRNA sono prodotti dall’elaborazione intracellulare di trascrizioni RNA hairpin più lunghe (riviste nelle referenze 17 e 18). Quest’ultimo approccio si basa principalmente sulla sintesi nucleare di RNA a forcina corta (shRNA), che vengono trasportati nel citoplasma attraverso la via di esportazione dei miRNA e vengono trasformati in siRNA da Dicer. Mentre l’uso diretto di effettori sintetici di siRNA è semplice e di solito risulta in un potente silenziamento genico, l’effetto è transitorio. I farmaci RNAi basati sul DNA, d’altra parte, hanno il potenziale di essere introdotti in modo stabile quando vengono usati in un contesto di terapia genica, permettendo, in linea di principio, un singolo trattamento di geni shRNA consegnati da vettori virali.
Le prime applicazioni cliniche di RNAi sono state dirette al trattamento della degenerazione maculare legata all’età (AMD), che causa ogni anno cecità o visione limitata in milioni di adulti (19,20). Terapie basate su RNAi sono attualmente in fase di sviluppo anche per le infezioni virali, tra cui il virus dell’immunodeficienza umana (HIV), i virus dell’epatite B e C (HBV e HCV) e il virus respiratorio sinciziale (RSV) (21). Anche le strategie per il trattamento delle malattie neurodegenerative e dei tumori sono ben avviate.
Anche se gli studi in vivo di successo hanno dimostrato la potenziale efficacia delle terapie basate sull’RNAi, altri studi hanno illustrato gli approcci specifici da evitare quando si adotta un meccanismo cellulare endogeno per un beneficio terapeutico. Gli effetti collaterali indesiderati hanno incluso l’attivazione dei recettori Toll-like (TLR) e le risposte dell’interferone di tipo 1, e la competizione con i componenti endogeni della via RNAi (22). Questi risultati indicano che, sebbene l’RNAi sia potenzialmente un meccanismo rivoluzionario per il trattamento delle malattie, è necessaria la dovuta cautela nell’interpretare i risultati degli abbattimenti dei target mediati dall’RNAi.
La sfida della consegna specifica di cellule o tessuti di siRNA è anche cruciale quando si studia l’utilità delle terapie basate sull’RNAi per una data malattia; varie strategie per la consegna non virale e virale dei trigger RNAi hanno dimostrato di essere efficaci nei loro rispettivi modelli di malattia. I vantaggi e gli svantaggi relativi dell’uso di siRNA sintetici rispetto agli shRNA espressi devono anche essere presi in considerazione quando si progettano terapie basate sull’RNAi per una particolare malattia.
I siRNA sintetizzati chimicamente sono comunemente sottoposti a screening per un efficace abbattimento di un gene bersaglio specifico. Per aumentare la stabilità del siRNA, vengono introdotte modifiche chimiche, come 2′-O-metilpurine o 2′-fluoropirimidine (23). Quando inizialmente si progetta una molecola di siRNA, si usano abitualmente algoritmi computazionali che incorporano vari parametri, tra cui la stabilità del duplex del siRNA per una corretta selezione dei filamenti e le strutture secondarie dell’mRNA per l’accessibilità al sito bersaglio. Per migliorare la potenza di una risposta RNAi, i duplex di siRNA possono anche essere progettati per imitare i substrati per l’elaborazione Dicer (24). I siRNA più lunghi (ad esempio, 27-mers) sono incorporati nella fase di caricamento Dicer del percorso RNAi e possono facilitare l’attivazione di RISC. 27-mers sono progettati asimmetricamente per mostrare un 2 nt 3′ overhang su un’estremità e una regione smussata dall’altro (25), che guida Dicer elaborazione e la biogenesi del filamento guida corretta, dal momento che il dominio PAZ di Dicer riconosce l’estremità overhang. Inoltre, a causa dell’efficienza dei 27-meri nel mediare il silenziamento genico, una minore concentrazione di siRNA può mediare una potente risposta RNAi.
Dalla prospettiva delle applicazioni terapeutiche dell’RNAi, la preoccupazione più importante è la consegna dei siRNA al tessuto appropriato. Numerose pubblicazioni recenti hanno dimostrato che i siRNA possono essere consegnati sistematicamente a vari tessuti con conseguente abbattimento degli RNA target. L’iniezione endovenosa di siRNA per la consegna sistemica è realizzata attraverso la coniugazione di molecole di siRNA ad un gruppo di colesterolo o il confezionamento di siRNA in particelle liposomiali. La consegna sistemica utilizzando questi approcci è efficace per la consegna al fegato e al digiuno, ma potrebbe non essere appropriata per la consegna ad altri organi. In uno studio proof-of-concept, i siRNA che prendono di mira l’apolipoproteina B (APOB) sono stati utilizzati per modificare il metabolismo del colesterolo. Il gruppo idrossile 3′ sul filamento passeggero del siRNA è stato legato chimicamente a un gruppo di colesterolo, e questi siRNA coniugati hanno effettivamente abbattuto l’espressione genica del >50% nel fegato e del 70% nel digiuno (26).
Un altro approccio per la consegna sistemica prevede l’uso di bilayer lipidici specializzati chiamati particelle stabili acido nucleico-lipidi (SNALPs), che incorporano siRNA chimicamente modificati (27). I lipidi cationici e neutri comprendono il bilayer, insieme a un rivestimento esterno idrofilo di polietilenglicole (PEG). In uno studio, alle scimmie è stata somministrata una singola dose di SNALPs contenenti siRNA, che hanno abbassato i livelli di colesterolo per 11 giorni o più, con < il 10% di espressione APOB rimanente nel fegato di questo modello di primate non umano (28). Non sono state osservate tossicità degne di nota, suggerendo la potenziale utilità di questo metodo nella consegna sistemica.
Per l’efficacia in vivo delle molecole di siRNA, il dosaggio dei siRNA consegnati è una considerazione pratica, e la consegna selettiva dei siRNA a tessuti specifici potrebbe potenzialmente abbassare il dosaggio effettivo richiesto. Il targeting dei recettori della superficie cellulare è un approccio vantaggioso, in quanto abbasserebbe il dosaggio di siRNA e potenzialmente eviterebbe effetti fuori bersaglio dalla consegna di siRNA a tessuti irrilevanti. L’accoppiamento di siRNA ad aptamers o frammenti di anticorpi, o l’uso di nanoparticelle rivestite con ligandi specifici del recettore, permette la consegna specifica di carichi utili di siRNA a cellule e tessuti mirati. Per il targeting delle cellule infette da HIV, gli siRNA sono stati accoppiati a frammenti di anticorpi a catena pesante (Fabs) che riconoscono la glicoproteina gp120 dell’involucro dell’HIV. La protamina caricata positivamente è stata coniugata alle molecole Fab, e gli siRNA caricati negativamente hanno interagito elettrostaticamente con la protamina per formare un complesso Fab-siRNA. Questo approccio basato sull’anticorpo ha dimostrato > un abbattimento del 70% della proteina p24 dell’antigene gruppo-specifico (Gag) quando si puntavano i linfociti T in coltura infettati con l’HIV-1 (29). Un diverso approccio di targeting ha sfruttato un peptide del virus della rabbia, che si lega specificamente al recettore dell’acetilcolina. Quando questo peptide è stato coniugato con un peptide di poliarginina che lega i siRNA, la consegna dei siRNA al sistema nervoso centrale è stata realizzata, con conseguente inibizione di un’infezione virale di encefalite fatale (30).
Gli aptamers, che sono ligandi strutturati di RNA, possono essere progettati per legarsi specificamente ai recettori della superficie cellulare ed essere legati covalentemente ai siRNAs per la consegna specifica in vivo. Un metodo ha utilizzato aptamers che si legano all’antigene di membrana specifico della prostata (PSMA) espresso sulla superficie delle cellule del cancro alla prostata. Quando coniugati a siRNA, questi ibridi aptamer-siRNA hanno ridotto efficacemente la crescita tumorale nei topi (31). Un approccio simile utilizzando sia siRNA biotinilati che aptameri legati alla streptavidina, proteina legante la biotina, ha fatto uso di siRNA a 27-mer per indurre potentemente il silenziamento genico (32).
Coating nanoparticelle con ligandi specifici del tipo di cellula è un altro potente approccio per fornire sistematicamente molecole che inducono RNAi. In un importante studio proof-of-concept, i tumori del sarcoma di Ewing sono stati presi di mira in vivo con nanoparticelle rivestite con ligandi della transferrina (33). Queste nanoparticelle sono state costruite usando policazioni contenenti ciclodestrina (CDP) specificamente progettate per incorporare molecole di siRNA caricate negativamente. Per una maggiore stabilità e per prevenire l’aggregazione, i polimeri PEG sono stati attaccati alla superficie esterna utilizzando gruppi terminali adamantani. I ligandi della transferrina sono stati poi legati covalentemente alle catene adamantano-PEG, e il design delle nanoparticelle ha permesso l’auto-assemblaggio in nanoparticelle uniformi, di dimensioni ∼50 nanometriche. Gli siRNA incorporati nelle nanoparticelle hanno preso di mira il prodotto di fusione del gene Ews-Fli1 (Ewing sarcoma breakpoint region 1-flightless 1 homolog) e hanno dimostrato di inibire la formazione del tumore nei topi (33).
Raccomandazioni conclusive
In sintesi, la progressione dalla scoperta iniziale della RNAi alle sue applicazioni cliniche è stata sorprendente. La comprensione della biologia fondamentale della RNAi ha portato alle sue applicazioni diffuse nella ricerca di base e successivamente alle applicazioni per il trattamento delle malattie. Entro i prossimi anni dovremmo aspettarci di scoprire un maggior numero di regolazioni dell’espressione genica mediate da RNAi, e vedremo anche farmaci basati su RNAi approvati per l’uso nel trattamento delle malattie. Inoltre, l’RNAi ha dimostrato di essere un potente strumento per lo studio della funzione genica e ha aperto nuove aree di indagine di base. Nel prossimo futuro dovremmo vedere un continuo sviluppo nella nostra comprensione e applicazione di questo notevole meccanismo cellulare per la regolazione post-trascrizionale dell’espressione genica.
Riconoscimenti
Questo lavoro è stato sostenuto dal National Institutes of Health National Institute of Allergy and Infectious Diseases e dal NIH Heart Lung and Blood Institute a J.J.R. D.H.K. è sostenuto da una borsa di studio pre-dottorato City of Hope.
Dichiarazione di interessi concorrenti
Gli autori non dichiarano interessi concorrenti.
- 1. Almeida, R. e R.C. Allshire. 2005. RNA silencing e regolazione del genoma. Trends Cell Biol. 15:251-258.Crossref, Medline, CAS, Google Scholar
- 2. Zhang, H., F.A. Kolb, L. Jaskiewicz, E. Westhof, and W. Filipowicz. 2004. Modelli di centro di elaborazione singolo per Dicer umano e RNase III batterico. Cell 118:57-68.Crossref, Medline, CAS, Google Scholar
- 3. Lee, Y., I. Hur, S.Y. Park, Y.K. Kim, M.R. Suh, e V.N. Kim. 2006. Il ruolo di PACT nel percorso di silenziamento dell’RNA. EMBO J. 25:522-532.Crossref, Medline, CAS, Google Scholar
- 4. Meister, G., M. Landthaler, A. Patkaniowska, Y. Dorsett, G. Teng, e T. Tuschl. 2004. Argonaute2 umano media la scissione dell’RNA mirata da miRNA e siRNA. Mol. Cell 15:185-197.Crossref, Medline, CAS, Google Scholar
- 5. Liu, J., M.A. Carmell, F.V. Rivas, C.G. Marsden, J.M. Thomson, J.J. Song, S.M. Hammond, L. Joshua-Tor, et al. 2004. Argonaute2 è il motore catalitico dell’RNAi dei mammiferi. Science 305:1437-41.Crossref, Medline, CAS, Google Scholar
- 6. Tang, G. 2005. siRNA e miRNA: una visione dei RISC. Trends Biochem. Sci. 30:106-114.Crossref, Medline, CAS, Google Scholar
- 7. Schwarz, D.S., G. Hutvagner, T. Du, Z. Xu, N. Aronin, e P.D. Zamore. 2003. Asimmetria nell’assemblaggio del complesso enzimatico RNAi. Cell 115:199-208.Crossref, Medline, CAS, Google Scholar
- 8. Khvorova, A., A. Reynolds, e S.D. Jayasena. 2003. Funzionale siRNAs e miRNAs mostrano bias filo. Cell 115:209-216.Crossref, Medline, CAS, Google Scholar
- 9. Bartel, D.P. 2004. MicroRNA: genomica, biogenesi, meccanismo e funzione. Cell 116:281-297.Crossref, Medline, CAS, Google Scholar
- 10. Bartel, D.P. e C.Z. Chen. 2004. Micromanager dell’espressione genica: l’influenza potenzialmente diffusa dei microRNA dei metazoi. Nat. Rev. Genet. 5:396-400.Crossref, Medline, CAS, Google Scholar
- 11. Bagga, S., J. Bracht, S. Hunter, K. Massirer, J. Holtz, R. Eachus, e A.E. Pasquinelli. 2005. Regolazione da let-7 e lin-4 miRNAs risultati nella degradazione mRNA bersaglio. Cell 122:553-563.Crossref, Medline, CAS, Google Scholar
- 12. Leuschner, P.J., S.L. Ameres, S. Kueng, e J. Martinez. 2006. Cleavage del filamento passeggero siRNA durante l’assemblaggio RISC in cellule umane. EMBO Rep. 7:314-320.Crossref, Medline, CAS, Google Scholar
- 13. Gregory, R.I., T.P. Chendrimada, N. Cooch, e R. Shiekhattar. 2005. RISC umano accoppia la biogenesi dei microRNA e il silenziamento genico post-trascrizionale. Cell 123:631-640.Crossref, Medline, CAS, Google Scholar
- 14. Matranga, C., Y. Tomari, C. Shin, D.P. Bartel, e P.D. Zamore. 2005. Passenger-strand cleavage facilita l’assemblaggio di siRNA in complessi enzimatici RNAi contenenti Ago2. Cell 123:607-620.Crossref, Medline, CAS, Google Scholar
- 15. Lee, Y., C. Ahn, J. Han, H. Choi, J. Kim, J. Yim, J. Lee, P. Provost, et al. 2003. Il nucleare RNase III Drosha inizia microRNA elaborazione. Natura 425:415-419.Crossref, Medline, CAS, Google Scholar
- 16. Han, J., Y. Lee, K.H. Yeom, Y.K. Kim, H. Jin, e V.N. Kim. 2004. Il complesso Drosha-DGCR8 nell’elaborazione primaria del microRNA. Genes Dev. 18:3016-3027.Crossref, Medline, CAS, Google Scholar
- 17. Hannon, G.J. e J.J. Rossi. 2004. Sbloccare il potenziale del genoma umano con l’interferenza RNA. Nature 431:371-378.Crossref, Medline, CAS, Google Scholar
- 18. Scherer, L.J. e J.J. Rossi. 2003. Approcci per il knockdown sequenza-specifico di mRNA. Nat. Biotechnol. 21:1457-1465.Crossref, Medline, CAS, Google Scholar
- 19. Fattal, E. e A. Bochot. 2006. Consegna oculare di acidi nucleici: oligonucleotidi antisenso, aptamers e siRNA. Adv. Drug Deliv. Rev. 58:1203-1223.Crossref, Medline, CAS, Google Scholar
- 20. Tolentino, M. 2006. Tecnologia RNA di interferenza nel trattamento della CNV. Ophthalmol. Clin. North Am. 19:393-399 vi-vii.Medline, Google Scholar
- 21. Leonard, J.N. e D.V. Schaffer. 2006. Terapia RNAi antivirale: approcci emergenti per colpire un bersaglio mobile. Gene Ther. 13:532-540.Crossref, Medline, CAS, Google Scholar
- 22. Svoboda, P. 2007. Off-targeting e altri effetti non specifici di esperimenti RNAi in cellule di mammifero. Curr. Opin. Mol. Ther. 9:248-257.Medline, CAS, Google Scholar
- 23. Czauderna, F., M. Fechtner, S. Dames, H. Aygun, A. Klippel, G.J. Pronk, K. Giese, e J. Kaufmann. 2003. Variazioni strutturali e modifiche stabilizzanti di siRNA sintetici in cellule di mammifero. Nucleic Acids Res. 31:2705-2716.Crossref, Medline, CAS, Google Scholar
- 24. Kim, D.H., M.A. Behlke, S.D. Rose, M.S. Chang, S. Choi, e J.J. Rossi. 2005. Sintetico dsRNA Dicer substrati migliorare RNAi potenza ed efficacia. Nat. Biotechnol. 23:222-226.Crossref, Medline, CAS, Google Scholar
- 25. Amarzguioui, M., P. Lundberg, E. Cantin, J. Hagstrom, M.A. Behlke, e J.J. Rossi. 2006. Progettazione razionale e consegna in vitro e in vivo di Dicer substrato siRNA. Nat. Protocols 1:508-517.Crossref, Medline, CAS, Google Scholar
- 26. Soutschek, J., A. Akinc, B. Bramlage, K. Charisse, R. Constien, M. Donoghue, S. Elbashir, A. Geick, et al. 2004. Silenziamento terapeutico di un gene endogeno mediante somministrazione sistemica di siRNA modificati. Nature 432:173-178.Crossref, Medline, CAS, Google Scholar
- 27. Morrissey, D.V., J.A. Lockridge, L. Shaw, K. Blanchard, K. Jensen, W. Breen, K. Hartsough, L. Machemer, et al. 2005. Potente e persistente attività anti-HBV in vivo di siRNA chimicamente modificati. Nat. Biotechnol. 23:1002-1007.Crossref, Medline, CAS, Google Scholar
- 28. Zimmermann, T.S., A.C. Lee, A. Akinc, B. Bramlage, D. Bumcrot, M.N. Fedoruk, J. Harborth, J.A. Heyes, et al. 2006. RNAi-mediato silenziamento del gene in primati non umani. Nature 441:111-114.Crossref, Medline, CAS, Google Scholar
- 29. Song, E., P. Zhu, S.K. Lee, D. Chowdhury, S. Kussman, D.M. Dykxhoorn, Y. Feng, D. Palliser, et al. 2005. Consegna in vivo mediata dall’anticorpo di piccoli RNA interferenti attraverso i recettori della superficie cellulare. Nat. Biotechnol. 23:709-717.Crossref, Medline, CAS, Google Scholar
- 30. Kumar, P., H. Wu, J.L. McBride, K.E. Jung, M.H. Kim, B.L. Davidson, S.K. Lee, P. Shankar, et al. 2007. Consegna transvascolare di piccolo RNA interferente al sistema nervoso centrale. Natura 448:39-43.Crossref, Medline, CAS, Google Scholar
- 31. McNamara, J.O., II, E.R. Andrechek, Y. Wang, K.D. Viles, R.E. Rempel, E. Gilboa, B.A. Sullenger, e P.H. Giangrande. 2006. Cell type-specific consegna di siRNAs con aptamer-siRNA chimere. Nat. Biotechnol. 24:1005-1015.Crossref, Medline, CAS, Google Scholar
- 32. Chu, T.C., K.Y. Twu, A.D. Ellington, e M. Levy. 2006. Aptamer mediato consegna siRNA. Nucleic Acids Res. 34:e73.Crossref, Medline, Google Scholar
- 33. Hu-Lieskovan, S., J.D. Heidel, D.W. Bartlett, M.E. Davis, e T.J. Triche. 2005. Abbattimento sequenza-specifico di EWS-FLI1 da mirato, consegna nonvirale di piccolo RNA interferente inibisce la crescita del tumore in un modello murino di sarcoma di Ewing metastatico. Cancer Res. 65:8984-8992.Crossref, Medline, CAS, Google Scholar