Introdução
Interferência do RNAi (RNAi) é um mecanismo regulador da maioria das células eucarióticas que utiliza pequenas moléculas de RNA de dupla cadeia (dsRNA) como estímulos para o controlo directo da actividade genética dependente de homólogos (Figura 1) (1). Conhecidas como pequenos RNA interferentes (siRNA), estas ∼21-22 bp moléculas de dsRNA longo têm características 2 nt 3′ overhangs que permitem o seu reconhecimento pela maquinaria enzimática do RNAi, o que eventualmente leva à degradação homóloga dependente do mRNA alvo. Nas células de mamíferos, os siRNAs são produzidos a partir da clivagem de precursores de dsRNA mais longos pelo Dicer de endonuclease RNase III (2), ou podem ser sintetizados por métodos químicos ou bioquímicos. O dicer é complexado com proteínas de ligação ao RNA, a proteína de ligação ao TAR-RNA (TRBP), PACT, e Ago-2, que estão envolvidas na entrega de siRNAs ao complexo de silenciamento induzido pelo RNA (RISC) (3). Os componentes centrais do RISC são os membros da família Argonaute (Ago). Nos humanos existem oito membros desta família mas apenas o Ago-2 possui um domínio catalítico activo para a actividade de clivagem (4,5). Enquanto os siRNAs carregados em RISC são de dupla cadeia, os Ago-2 clivam e libertam o cordão “passageiro”, levando a uma forma activada de RISC com uma molécula “guia” de RNA de cadeia única que dirige a especificidade do reconhecimento do alvo por emparelhamento de base intermolecular (6). As regras que governam a selectividade da carga de cordão em RISC baseiam-se em estabilidades termodinâmicas diferenciais das extremidades dos siRNAs (7,8). A extremidade menos termodinamicamente estável é favorecida para ligação ao domínio PIWI de Ago-2.
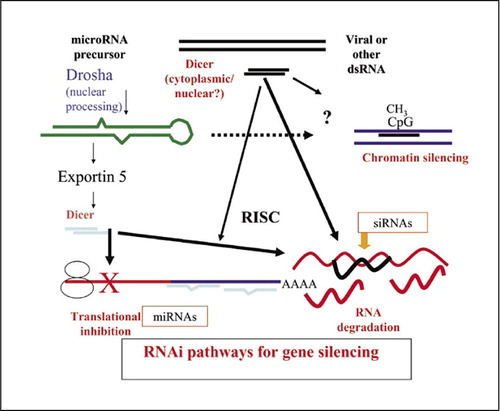
RNAi é multifacetado, e existem várias vias nas quais pequenos RNA de fio duplo (dsRNAs) regulam a expressão genética. A via do micro-RNA endógeno (miRNA) começa com os miRNAs primários transcritos por Pol II que são processados no núcleo para os prémiRNAs, exportados para o citoplasma, e processados novamente em miRNAs funcionais. A função primária dos miRNAs é inibir a tradução através do emparelhamento incompleto da base Watson-Crick para as regiões não traduzidas dos mRNAs alvo 3′. Alternativamente, pequenos RNAs interferentes (siRNAs) perfeitamente duplexados podem ser produzidos intracelularmente ou fornecidos exogenamente às células. A vertente guia é incorporada no complexo de silenciamento induzido por RNA (RISC), onde orienta a degradação específica da sequência da transcrição do alvo, independentemente do local onde ocorre o emparelhamento de base. As vias do miRNA e do siRNA são intercambiáveis, e os determinantes importantes são as posições dentro da mensagem e a extensão do emparelhamento de base com as transcrições alvo. Os siRNAs também podem desencadear o silenciamento transcricional de genes através de interacções com a cromatina, onde guiam a história e a metilação do ADN levando à cromatina inactiva.
MicroRNAs
Um importante braço do RNAi envolve os microRNAs (miRNAs). Estes são dúplexos endógenos que regulam a expressão genética pós-transcrição através de complexação com RISC e ligação às regiões não traduzidas (UTRs) de sequências alvo através de pequenos trechos de homologia, denominadas “sequências de sementes” (9,10). O principal mecanismo de acção dos miRNAs é a repressão translacional, embora esta possa ser acompanhada pela degradação da mensagem (11). Os duplexes miRNA possuem emparelhamento de base Watson-Crick incompleto, e a vertente antisense não pode ser escolhida por clivagem da vertente passageiro como é para os siRNAs; por conseguinte, a vertente antisense deve ser escolhida por um mecanismo alternativo (12-14). os miRNAs são substratos endógenos para a maquinaria do RNAi. São inicialmente expressos como transcrições primárias longas (pri-miRNAs), que são processados dentro do núcleo em grampos de cabelo de 60-70 bp pelo complexo do microprocessador, consistindo de Drosha e DGCR8 (15,16) em prémiRNAs. Os prémiRNAs são processados no citoplasma por Dicer e um dos dois fios é carregado no RISC, presumivelmente através da interacção com uma das proteínas acessórias de Dicer (3). É importante notar que é possível explorar esta via de silenciamento de genes nativos para a regulação do(s) gene(s) escolhido(s). Se o efeito siRNA for entregue à célula, “activará” o RISC, resultando num silenciamento potente e específico do mRNA visado. Devido à potência e selectividade do RNAi, tornou-se a metodologia de escolha para silenciar a expressão genética específica em células de mamíferos.
RNAi como Abordagem Terapêutica para o Tratamento da Doença
Controlo dos genes associados à doença torna o RNAi uma escolha atractiva para a terapêutica futura. Basicamente, cada doença humana causada por actividade de um ou poucos genes deve ser passível de intervenção com base no RNAi. Esta lista inclui cancro, doenças auto-imunes, doenças genéticas dominantes, e infecções virais. O RNAi pode ser desencadeado por duas vias diferentes: (i) uma abordagem baseada no RNAi, em que os siRNAs efetores sintéticos são entregues por vários portadores a células alvo como duplex de 21 bases pré-formadas; ou (ii) através de estratégias baseadas no ADN, em que os siRNA efetores são produzidos por processamento intracelular de transcrições mais longas do fio de cabelo de RNAi (revistas nas Referências 17 e 18). Esta última abordagem é baseada principalmente na síntese nuclear de RNAs de citoplasma de cadeia curta (shRNAs), que são transportados para o citoplasma através da via de exportação do miRNA e são processados em siRNAs por Dicer. Embora a utilização directa de efectores de siRNA sintéticos seja simples e normalmente resulte num potente silenciamento genético, o efeito é transitório. Os medicamentos RNAi baseados em ADN, por outro lado, têm o potencial de serem introduzidos de forma estável quando utilizados num ambiente de terapia genética, permitindo, em princípio, um tratamento único dos genes do shRNA transmitido por vectores virais.
p>As primeiras aplicações clínicas do RNAi têm sido dirigidas ao tratamento da degeneração macular relacionada com a idade (DMRI), que causa cegueira ou visão limitada em milhões de adultos anualmente (19,20). As terapias baseadas no RNAi também estão actualmente a ser desenvolvidas para a infecção viral, incluindo o vírus da imunodeficiência humana (HIV), os vírus da hepatite B e C (HBV e HCV), e o vírus sincicial respiratório (RSV) (21). As estratégias para o tratamento de doenças neurodegenerativas e cancros estão também bem encaminhadas.p>Embora estudos in vivo bem sucedidos tenham demonstrado a eficácia potencial das terapias baseadas no RNAi, outros estudos ilustraram abordagens específicas a evitar quando se adopta um mecanismo celular endógeno para benefício terapêutico. Os efeitos secundários indesejados incluíram a activação de receptores tipo Toll-like (TLRs) e respostas de interferão de tipo 1, e competição com os componentes endógenos da via do RNAi (22). Estas descobertas indicam que embora o RNAi seja potencialmente um mecanismo revolucionário para o tratamento de doenças, é necessária a devida cautela ao interpretar os resultados de nocaute de alvo mediado por RNAi.
O desafio da administração de siRNAs específicos de células ou tecidos é também crucial ao investigar a utilidade das terapias baseadas em RNAi para uma dada doença; várias estratégias para a administração não viral e viral de desencadeadores de RNAi têm demonstrado ser eficazes nos seus respectivos modelos de doença. As vantagens e desvantagens relativas da utilização de siRNAs sintéticos versus shRNAs expressos devem também ser tidas em consideração ao conceber terapias baseadas em RNAi para uma determinada doença.
SiRNAs sintetizados quimicamente são normalmente analisados para a eliminação eficaz de um gene alvo específico. Para aumentar a estabilidade do siRNA, são introduzidas modificações químicas, tais como 2′-O-metilpurinas ou 2′-fluoropyrimidinas (23). Na concepção inicial de uma molécula de siRNA, são rotineiramente utilizados algoritmos computacionais que incorporam vários parâmetros, incluindo estabilidades finais duplex de siRNA para uma selecção adequada de cordão e estruturas secundárias de mRNA para acessibilidade do local alvo. Para melhorar a potência de uma resposta de RNAi, os duplex de siRNA também podem ser concebidos para imitar substratos para processamento de Dicer (24). Os siRNAs mais longos (por exemplo, 27 mers) são incorporados na etapa de carregamento de Dicer da via de RNAi e podem facilitar a activação de RISC. Os 27-mers são concebidos assimetricamente para exibir uma saliência de 2 nt 3′ numa extremidade e uma região romba na outra (25), que orienta o processamento de Dicer e a biogénese da vertente guia adequada, uma vez que o domínio PAZ de Dicer reconhece a extremidade da saliência. Além disso, devido à eficiência de 27 homens no silenciamento de genes mediadores, uma menor concentração de siRNAs pode mediar uma resposta potente de RNAi.
Da perspectiva das aplicações terapêuticas do RNAi, a preocupação mais importante é a entrega dos siRNAs ao tecido apropriado. Numerosas publicações recentes mostraram que os siRNAs podem ser sistematicamente entregues a vários tecidos com a consequente eliminação dos RNAi alvo. A injecção intravenosa de siRNAs para entrega sistémica é realizada através da conjugação de moléculas de siRNA a um grupo de colesterol ou da embalagem de siRNAs em partículas lipossómicas. A entrega sistémica utilizando estas abordagens é eficaz para a entrega ao fígado e jejuno, mas pode não ser apropriada para a entrega a outros órgãos. Num estudo de prova de conceito, os siRNAs visando a apolipoproteína B (APOB) foram utilizados para modificar o metabolismo do colesterol. O grupo 3′ hydroxyl na vertente de passageiros do siRNA foi quimicamente ligado a um grupo de colesterol, e estes siRNAs conjugados derrubaram efectivamente a expressão genética por >50% no fígado e 70% no jejuno (26).
p> Outra abordagem para a entrega sistémica envolve o uso de bilayers lipídicos especializados denominados partículas nucleico-lípidas estáveis (SNALPs), que incorporam siRNAs quimicamente modificados (27). Os lípidos catiónicos e neutros compreendem o bocal, juntamente com um revestimento hidrofílico externo de polietilenoglicol (PEG). Num estudo, foi administrada aos macacos uma dose única de siRNA contendo SNALPs, que baixou os níveis de colesterol durante 11 dias ou mais, com <10% da expressão APOB remanescente no fígado deste modelo de primata não-humano (28). Não foram observadas toxicidades perceptíveis, sugerindo a potencial utilidade deste método na entrega sistémica.
Para a eficácia in vivo das moléculas de siRNA, a dosagem de siRNAs entregues é uma consideração prática, e a entrega selectiva de siRNAs a tecidos específicos reduziria potencialmente a dose efectiva necessária. O alvo dos receptores de superfície celular é uma abordagem vantajosa, uma vez que baixaria a dosagem de siRNA e potencialmente evitaria efeitos fora do alvo da entrega de siRNA a tecidos irrelevantes. O acoplamento de siRNA aos aptamers ou fragmentos de anticorpos, ou a utilização de nanopartículas revestidas com ligandos específicos do receptor, permite a entrega específica de cargas úteis de siRNA a células e tecidos alvo. Para a detecção de células infectadas com VIH, os siRNAs foram acoplados a fragmentos de anticorpos de cadeia pesada (Fabs) que reconhecem a glicoproteína gp120 do envelope do VIH. A protamina com carga positiva foi conjugada a moléculas Fab, e os siRNAs com carga negativa interagiram electrostaticamente com a protamina para formar um complexo Fab-siRNA. Esta abordagem baseada em anticorpos demonstrou >70% de derrubamento da proteína antigénica específica do grupo p24 (Gag) ao visar linfócitos T de cultura infectados com HIV-1 (29). Uma abordagem diferente da focalização tirou partido de um peptídeo do vírus da raiva, que se liga especificamente ao receptor de acetilcolina. Quando este peptídeo foi conjugado a um peptídeo de poliarginina que liga os siRNAs, a entrega de siRNAs ao sistema nervoso central foi realizada, resultando na inibição de uma infecção viral por encefalite fatal (30).
Aptamers, que são ligandos estruturados de RNA, podem ser concebidos para se ligarem especificamente aos receptores de superfície celular e serem ligados covalentemente aos siRNAs para entrega específica in vivo. Um método utilizou aptamers que se ligam ao antigénio de membrana específico da próstata (PSMA) expresso na superfície das células cancerosas da próstata. Quando conjugados com siRNAs, estes aptamer-siRNA híbridos reduziram efectivamente o crescimento tumoral em ratos (31). Uma abordagem semelhante utilizando tanto siRNAs biotinilados como aptamers ligados à estreptavidina proteica de ligação à biotina fez uso de siRNAs de 27 mers para induzir potentemente o silenciamento genético (32).
Coating nanoparticles with cell type-specific ligands is another powerful approach to deliver sistemically RNAi-inducing molecules. Num importante estudo de prova de conceito, os tumores do sarcoma de Ewing foram alvo in vivo com nanopartículas revestidas com ligantes de transferrina (33). Estas nanopartículas foram construídas utilizando policações contendo ciclodextrina (CDPs) especificamente concebidas para incorporar moléculas de siRNA carregadas negativamente. Para maior estabilidade e para evitar agregação, os polímeros de PEG foram ligados à superfície externa utilizando grupos terminais adamantanosos. Os ligandos de transferência foram então covalentemente ligados às cadeias adamantano-PEG, e o desenho de nanopartículas permitiu a auto-montagem em nanopartículas uniformes, ∼50 nanopartículas do tamanho de nanómetro. Os siRNAs incorporados em nanopartículas visavam o produto de fusão genética Ews-Fli1 (Ewing sarcoma breakpoint region 1-flightless 1 homologado) e mostraram inibir a formação de tumor l em ratos (33).
Comentários finais
Em resumo, a progressão desde a descoberta inicial do RNAi até às suas aplicações clínicas tem sido espantosa. A compreensão da biologia fundamental do RNAi levou às suas aplicações generalizadas na investigação básica e, subsequentemente, em aplicações para o tratamento de doenças. Nos próximos anos devemos esperar desvendar mais regulamentação da expressão genética mediada pelo RNAi, e veremos também os medicamentos baseados no RNAi aprovados para uso no tratamento de doenças. Além disso, o RNAi provou ser uma ferramenta poderosa para o estudo da função do gene e abriu novas áreas de investigação básica. Num futuro próximo devemos ver um desenvolvimento contínuo na nossa compreensão e aplicação deste notável mecanismo celular de regulação pós-transcricional da expressão genética.
Agradecimentos
Este trabalho foi apoiado pelo National Institutes of Health National Institute of Allergy and Infectious Diseases e pelo NIH Heart Lung and Blood Institute to J.J.R. D.H.K. é apoiado por uma bolsa de pré-doutoramento da Cidade da Esperança.
Declaração de interesses concorrentes
Os autores declaram não haver interesses concorrentes.
- 1. Almeida, R. e R.C. Allshire. 2005. Silenciamento do RNA e regulação do genoma. Trends Cell Biol. 15:251-258.Crossref, Medline, CAS, Google Scholar
- 2. Zhang, H., F.A. Kolb, L. Jaskiewicz, E. Westhof, e W. Filipowicz. 2004. Modelos de centro de processamento único para Dicer humano e RNase III bacteriano. Célula 118:57-68.Crossref, Medline, CAS, Google Scholar
- 3. Lee, Y., I. Hur, S.Y. Park, Y.K. Kim, M.R. Suh, e V.N. Kim. 2006. O papel do PACT no caminho de silenciamento do ARN. EMBO J. 25:522-532.Crossref, Medline, CAS, Google Scholar
- 5. Liu, J., M.A. Carmell, F.V. Rivas, C.G. Marsden, J.M. Thomson, J.J. Song, S.M. Hammond, L. Joshua-Tor, et al. 2004. Argonaute2 é o motor catalítico do RNAi de mamíferos. Science 305:1437-41.Crossref, Medline, CAS, Google Scholar
- 6. Tang, G. 2005. siRNA e miRNA: uma visão sobre RISCs. Tendências Biochem. Sci. 30:106-114.Crossref, Medline, CAS, Google Scholar
- 7. Schwarz, D.S., G. Hutvagner, T. Du, Z. Xu, N. Aronin, e P.D. Zamore. 2003. Assimetria na montagem do complexo enzimático RNAi. Cell 115:199-208.Crossref, Medline, CAS, Google Scholar
- 8. Khvorova, A., A. Reynolds, e S.D. Jayasena. 2003. SiRNAs funcionais e miRNAs exibem polarização de fios. Cell 115:209-216.Crossref, Medline, CAS, Google Scholar
- 10. Bartel, D.P. e C.Z. Chen. 2004. Micromanagers of gene expression: a influência potencialmente generalizada dos microRNAs metazoários. Nat. Rev. Genet. 5:396-400.Crossref, Medline, CAS, Google Scholar
- 11. Bagga, S., J. Bracht, S. Hunter, K. Massirer, J. Holtz, R. Eachus, e A.E. Pasquinelli. 2005. A regulação por let-7 e lin-4 miRNAs resulta na degradação do mRNA alvo. Cell 122:553-563.Crossref, Medline, CAS, Google Scholar
- 12. Leuschner, P.J., S.L. Ameres, S. Kueng, e J. Martinez. 2006. Limpeza do cordão de passageiros do siRNA durante a montagem do RISC em células humanas. EMBO Rep. 7:314-320.Crossref, Medline, CAS, Google Scholar
- 13. Gregory, R.I., T.P. Chendrimada, N. Cooch, e R. Shiekhattar. 2005. O RISC humano associa microRNA biogénese e silenciamento genético pós-transcritivo. Célula 123:631-640.Crossref, Medline, CAS, Google Scholar
- 14. Matranga, C., Y. Tomari, C. Shin, D.P. Bartel, e P.D. Zamore. 2005. A clivagem do fio do passageiro facilita a montagem do siRNA em complexos de enzimas RNAi contendo Ago2. Célula 123:607-620.Crossref, Medline, CAS, Google Scholar
- 15. Lee, Y., C. Ahn, J. Han, H. Choi, J. Kim, J. Yim, J. Lee, P. Provost, et al. 2003. O RNase III Drosha nuclear inicia o processamento de microRNA. Nature 425:415-419.Crossref, Medline, CAS, Google Scholar
- 16. Han, J., Y. Lee, K.H. Yeom, Y.K. Kim, H. Jin, e V.N. Kim. 2004. O complexo Drosha-DGCR8 no processamento primário de microRNA. Genes Dev. 18:3016-3027.Crossref, Medline, CAS, Google Scholar
- 17. Hannon, G.J. e J.J. Rossi. 2004. Desbloqueando o potencial do genoma humano com interferência do RNA. Nature 431:371-378.Crossref, Medline, CAS, Google Scholar
- 18. Scherer, L.J. e J.J. Rossi. 2003. Abordagens para o derrubamento específico da sequência de mRNA. Nat. Biotechnol. 21:1457-1465.Crossref, Medline, CAS, Google Scholar
- 19. Fattal, E. e A. Bochot. 2006. Entrega ocular de ácidos nucleicos: oligonucleótidos antisensos, aptamers e siRNA. Adv. Deliv. de drogas. Rev. 58:1203-1223.Crossref, Medline, CAS, Google Scholar
- 21. Leonard, J.N. e D.V. Schaffer. 2006. Antiviral RNAi therapy: abordagens emergentes para atingir um alvo em movimento. Gene Ther. 13:532-540.Crossref, Medline, CAS, Google Scholar
- 22. Svoboda, P. 2007. Off-targeting e outros efeitos não específicos das experiências de RNAi em células de mamíferos. Moeda. Opinião. Mol. Ther. 9:248-257.Medline, CAS, Google Scholar
- 23. Czauderna, F., M. Fechtner, S. Dames, H. Aygun, A. Klippel, G. J. Pronk, K. Giese, e J. Kaufmann. 2003. Variações estruturais e modificações estabilizadoras de siRNAs sintéticos em células de mamíferos. Ácidos Nucleicos Res. 31:2705-2716.Crossref, Medline, CAS, Google Scholar
- 24. Kim, D.H., M.A. Behlke, S.D. Rose, M.S. Chang, S. Choi, e J.J. Rossi. 2005. Os substratos sintéticos de dsRNA Dicer aumentam a potência e eficácia do RNAi. Nat. Biotechnol. 23:222-226.Crossref, Medline, CAS, Google Scholar
- 25. Amarzguioui, M., P. Lundberg, E. Cantin, J. Hagstrom, M.A. Behlke, e J.J. Rossi. 2006. Concepção racional e entrega in vitro e in vivo de SiRNA de substrato Dicer. Nat. Protocolos 1:508-517.Crossref, Medline, CAS, Google Scholar
- 26. Soutschek, J., A. Akinc, B. Bramlage, K. Charisse, R. Constien, M. Donoghue, S. Elbashir, A. Geick, et al. 2004. Silenciamento terapêutico de um gene endógeno através da administração sistémica de siRNAs modificados. Nature 432:173-178.Crossref, Medline, CAS, Google Scholar
- 28. Zimmermann, T.S., A.C. Lee, A. Akinc, B. Bramlage, D. Bumcrot, M.N. Fedoruk, J. Harborth, J.A. Heyes, et al. 2006. Silenciamento genético mediado por RNAi em primatas não humanos. Nature 441:111-114.Crossref, Medline, CAS, Google Scholar
- 29. Song, E., P. Zhu, S.K. Lee, D. Chowdhury, S. Kussman, D.M. Dykxhoorn, Y. Feng, D. Palliser, et al. 2005. Anticorpo mediado in vivo de pequenos RNAs interferentes através de receptores de superfície celular. Nat. Biotechnol. 23:709-717.Crossref, Medline, CAS, Google Scholar
- 30. Kumar, P., H. Wu, J.L. McBride, K.E. Jung, M.H. Kim, B.L. Davidson, S.K. Lee, P. Shankar, et al. 2007. Entrega transvascular de pequeno ARN interferente ao sistema nervoso central. Nature 448:39-43.Crossref, Medline, CAS, Google Scholar
- 31. McNamara, J.O., II, E.R. Andrechek, Y. Wang, K.D. Viles, R.E. Rempel, E. Gilboa, B.A. Sullenger, e P.H. Giangrande. 2006. Entrega de siRNAs com quimeras aptamer-siRNA de tipo celular específico. Nat. Biotechnol. 24:1005-1015.Crossref, Medline, CAS, Google Scholar
- 32. Chu, T.C., K.Y. Twu, A.D. Ellington, e M. Levy. 2006. Aptamer mediou a entrega do siRNA. Ácidos nucleicos Res. 34:e73.Crossref, Medline, Google Scholar
- 33. Hu-Lieskovan, S., J.D. Heidel, D.W. Bartlett, M.E. Davis, e T.J. Triche. 2005. O derrube de EWS-FLI1 por meio de uma entrega não viral de RNA com pequena interferência inibe o crescimento tumoral num modelo murino de sarcoma metastático de Ewing. Cancer Res. 65:8984-8992.Crossref, Medline, CAS, Google Scholar
li>4. Meister, G., M. Landthaler, A. Patkaniowska, Y. Dorsett, G. Teng, e T. Tuschl. 2004. Argonaute2 humano mede a clivagem do ARN visado por miRNAs e siRNAs. Mol. Cell 15:185-197.Crossref, Medline, CAS, Google Scholar
li>9. Bartel, D.P. 2004. MicroRNAs: genómica, biogénese, mecanismo, e função. Célula 116:281-297.Crossref, Medline, CAS, Google Scholar
li>20. Tolentino, M. 2006. Interferência da tecnologia do RNA no tratamento do CNV. Ophthalmol. Clin. Am. Norte. 19:393-399 vi-vii.Medline, Google Scholar
li>27. Morrissey, D.V., J.A. Lockridge, L. Shaw, K. Blanchard, K. Jensen, W. Breen, K. Hartsough, L. Machemer, et al. 2005. Potente e persistente actividade in vivo anti-HBV de siRNAs quimicamente modificados. Nat. Biotechnol. 23:1002-1007.Crossref, Medline, CAS, Google Scholar