Inleiding
RNA-interferentie (RNAi) is een reguleringsmechanisme van de meeste eukaryote cellen dat gebruik maakt van kleine dubbelstrengs RNA (dsRNA)-moleculen als triggers voor een directe homologie-afhankelijke controle van de genactiviteit (figuur 1) (1). Bekend als kleine interfererende RNA’s (siRNA), deze ∼21-22 bp lange dsRNA moleculen hebben karakteristieke 2 nt 3′ overhangs die hen in staat stellen te worden herkend door de enzymatische machinerie van RNAi, die uiteindelijk leidt tot homologie-afhankelijke afbraak van het doel-mRNA. In zoogdiercellen worden siRNA’s geproduceerd door splitsing van langere dsRNA precursors door het RNase III endonucle Dicer (2), of zij kunnen worden gesynthetiseerd door chemische of biochemische methoden. Dicer is gecomplexeerd met RNA-bindende eiwitten, het TAR-RNA-bindende eiwit (TRBP), PACT, en Ago-2, die betrokken zijn bij de overdracht van siRNA’s aan het RNA-geïnduceerde silencing complex (RISC) (3). De kerncomponenten van het RISC zijn de leden van de Argonaute-familie (Ago). Bij de mens zijn er acht leden van deze familie, maar alleen Ago-2 bezit een actief katalytisch domein voor splitsingsactiviteit (4,5). Terwijl siRNA’s die in het RISC worden geladen dubbelstrengs zijn, klieft Ago-2 en laat de “passagier”-streng los, wat leidt tot een geactiveerde vorm van het RISC met een enkelstrengs “gids”-RNA-molecuul dat de specificiteit van de doelherkenning regisseert door intermoleculaire baseparing (6). De regels voor de selectiviteit van de streng die in het RISC wordt geladen, zijn gebaseerd op de verschillende thermodynamische stabiliteit van de uiteinden van de siRNA’s (7,8). Het minder thermodynamisch stabiele uiteinde krijgt de voorkeur voor binding aan het PIWI-domein van Ago-2.
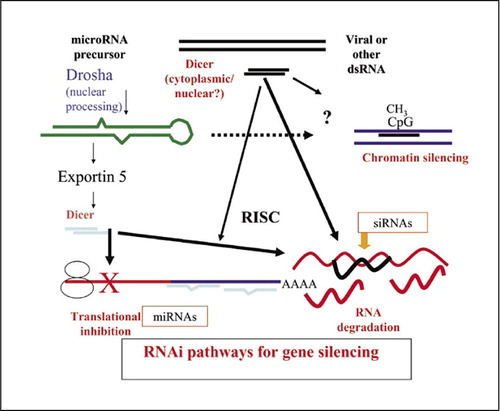
RNAi is veelzijdig, en er zijn verschillende routes waarin kleine dubbelstrengs RNA’s (dsRNA’s) de genexpressie reguleren. De endogene micro-RNA (miRNA) route begint met door Pol II getranscribeerde primaire miRNA’s die in de kern worden verwerkt tot pre-miRNA’s, naar het cytoplasma worden geëxporteerd, en daar opnieuw worden verwerkt tot functionele miRNA’s. De voornaamste functie van miRNA’s is het remmen van de translatie via onvolledige Watson-Crick baseparing aan de 3′-onvertaalde regio’s van doel-mRNA’s. Als alternatief kunnen perfect gedupliceerde kleine interfererende RNA’s (siRNA’s) intracellulair worden geproduceerd of exogeen aan cellen worden toegediend. De gidsstreng wordt opgenomen in het RNA-geïnduceerd silencing complex (RISC), waar hij de sequentiespecifieke afbraak van het doeltranscript stuurt, ongeacht waar de basenparing plaatsvindt. De miRNA- en siRNA-pathways zijn onderling verwisselbaar, en de belangrijke determinanten zijn de posities binnen de boodschap en de mate van basenparing met de doeltranscripten. siRNA’s kunnen ook transcriptionele gene silencing in gang zetten via interacties met chromatine, waarbij ze histon- en DNA-methylering sturen, wat leidt tot inactief chromatine.
MicroRNA’s
Een belangrijk onderdeel van RNAi zijn de microRNA’s (miRNA’s). Dit zijn endogene duplexen die posttranscriptioneel genexpressie reguleren door complexvorming met RISC en binding aan de 3′ untranslated regions (UTR’s) van doelsequenties via korte stukken homologie, de zogenaamde “zaadsequenties” (9,10). Het voornaamste werkingsmechanisme van miRNA’s is translatorische repressie, hoewel dit gepaard kan gaan met degradatie van de boodschap (11). De miRNA duplexen bezitten een onvolledige Watson-Crick basenparing, en de antisense streng kan niet worden gekozen door splitsing van de passagestreng zoals bij siRNAs het geval is; daarom moet de antisense streng door een alternatief mechanisme worden gekozen (12-14). miRNAs zijn endogene substraten voor de RNAi-machinerie. Zij komen aanvankelijk tot expressie als lange primaire transcripten (pri-miRNA’s), die in de kern door het Microprocessorcomplex, bestaande uit Drosha en DGCR8 (15,16), tot 60-70 bp haarspelden worden verwerkt tot pre-miRNA’s. De pre-miRNA’s worden door het Microprocessorcomplex in de kern verwerkt tot 60-70 bp haarspelden. De pre-miRNA’s worden in het cytoplasma verder verwerkt door Dicer en een van de twee strengen wordt in het RISC geladen, vermoedelijk via interactie met een van de Dicer accessoire-eiwitten (3). Belangrijk is dat het mogelijk is deze inheemse “gene silencing pathway” te benutten voor de regulering van gen(en) naar keuze. Als de siRNA-effector aan de cel wordt geleverd, wordt RISC “geactiveerd”, hetgeen resulteert in een krachtig en specifiek stilleggen van het beoogde mRNA. Door de kracht en selectiviteit van RNAi is het de methode bij uitstek geworden om specifieke genexpressie in zoogdiercellen tot zwijgen te brengen.
RNAi als therapeutische benadering voor de behandeling van ziekten
Beperking van met ziekten geassocieerde genen maakt RNAi een aantrekkelijke keuze voor toekomstige therapeutica. In principe zou elke menselijke ziekte, veroorzaakt door de activiteit van één of enkele genen, vatbaar moeten zijn voor RNAi-gebaseerde interventie. Deze lijst omvat kanker, auto-immuunziekten, dominante genetische aandoeningen, en virale infecties. RNAi kan op twee verschillende manieren in gang worden gezet: (i) een op RNA gebaseerde aanpak waarbij synthetische effector siRNA’s worden geleverd door verschillende dragers aan doelcellen als voorgevormde 21 base duplexen, of (ii) via DNA-gebaseerde strategieën waarbij de siRNA effectoren worden geproduceerd door intracellulaire verwerking van langere RNA hairpin transcripten (besproken in de referenties 17 en 18). Deze laatste aanpak is hoofdzakelijk gebaseerd op nucleaire synthese van korte haarspeld-RNA’s (shRNA’s), die naar het cytoplasma worden getransporteerd via de miRNA-exportroute en door Dicer tot siRNA’s worden verwerkt. Hoewel het directe gebruik van synthetische siRNA-effectoren eenvoudig is en gewoonlijk resulteert in een krachtige onderdrukking van het gen, is het effect van voorbijgaande aard. DNA-gebaseerde RNAi-geneesmiddelen, aan de andere kant, hebben het potentieel om stabiel te worden geïntroduceerd bij gebruik in een gentherapie setting, waardoor, in principe, een enkele behandeling van virale vector-geleverde shRNA genen.
De eerste klinische toepassingen van RNAi zijn gericht op de behandeling van leeftijdsgebonden maculaire degeneratie (AMD), die blindheid of beperkt gezichtsvermogen veroorzaakt bij miljoenen volwassenen per jaar (19,20). Therapieën op basis van RNAi worden momenteel ook ontwikkeld voor virale infecties, waaronder het humane immunodeficiëntievirus (HIV), hepatitis B- en C-virussen (HBV en HCV), en respiratoir syncytieel virus (RSV) (21). Strategieën voor de behandeling van neurodegeneratieve ziekten en kanker zijn ook in volle gang.
Hoewel succesvolle in vivo studies de potentiële effectiviteit van RNAi-gebaseerde therapieën hebben aangetoond, hebben andere studies specifieke benaderingen geïllustreerd die moeten worden vermeden bij het overnemen van een endogeen cellulair mechanisme voor therapeutisch voordeel. Ongewenste neveneffecten zijn onder meer activering van Toll-like receptoren (TLR’s) en type 1 interferon reacties, en concurrentie met de endogene RNAi pathway componenten (22). Deze bevindingen geven aan dat, hoewel RNAi is potentieel een revolutionair mechanisme voor de behandeling van ziekte, de nodige voorzichtigheid is geboden bij de interpretatie van de resultaten van RNAi-gemedieerde doel knockdowns.
De uitdaging van cel-of weefselspecifieke levering van siRNA’s is ook van cruciaal belang bij het onderzoeken van het nut van RNAi-gebaseerde therapieën voor een bepaalde ziekte; verschillende strategieën voor niet-virale en virale levering van RNAi triggers hebben aangetoond effectief te zijn in hun respectievelijke ziektemodellen. De relatieve voor- en nadelen van het gebruik van synthetische siRNA’s versus tot expressie gebrachte shRNA’s moeten ook in aanmerking worden genomen bij het ontwerpen van RNAi-gebaseerde therapieën voor een bepaalde ziekte.
Chemisch gesynthetiseerde siRNA’s worden vaak gescreend voor effectieve knockdown van een specifiek doelgen. Om de stabiliteit van siRNA te verhogen, worden chemische modificaties geïntroduceerd, zoals 2′-O-methylpurines of 2′-fluoropyrimidines (23). Bij het oorspronkelijke ontwerp van een siRNA molecuul, zijn computationele algoritmen routinematig gebruikt die verschillende parameters, met inbegrip van siRNA duplex einde structuren voor de juiste streng selectie en mRNA secundaire structuren voor doel site toegankelijkheid op te nemen. Om de potentie van een RNAi-respons te verbeteren, kunnen siRNA duplexen ook worden ontworpen om na te bootsen substraten voor Dicer verwerking (24). Langere siRNA’s (bijvoorbeeld 27-mers) worden opgenomen in de Dicer laden stap van de RNAi-route en kan de activering van RISC te vergemakkelijken. 27-mers zijn asymmetrisch ontworpen om een 2 nt 3′ overhang vertonen aan de ene kant en een stompe regio aan de andere kant (25), die Dicer verwerking en biogenese van de juiste gids streng leidt, omdat het PAZ domein van Dicer herkent de overhang einde. Bovendien, vanwege de efficiëntie van 27-mers in het bemiddelen gen silencing, kan een lagere concentratie van siRNAs mediëren een krachtige RNAi respons.
Vanuit het perspectief van therapeutische toepassingen van RNAi, de belangrijkste zorg is de levering van de siRNAs aan het juiste weefsel. Talrijke recente publicaties hebben aangetoond dat siRNA’s systemisch kunnen worden geleverd aan verschillende weefsels met als gevolg knockdown van doel RNA’s. Intraveneuze injectie van siRNA’s voor systemische toediening wordt bereikt door conjugatie van siRNA-moleculen aan een cholesterolgroep of de verpakking van siRNA’s in liposomale deeltjes. Systemische toediening met deze benaderingen is doeltreffend voor toediening aan de lever en het jejunum, maar is wellicht niet geschikt voor toediening aan andere organen. In een proof-of-conceptstudie werden siRNA’s gericht tegen apolipoproteïne B (APOB) gebruikt om het cholesterolmetabolisme te wijzigen. De 3′ hydroxylgroep op de siRNA-passagierstreng werd chemisch gekoppeld aan een cholesterolgroep, en deze geconjugeerde siRNA’s schakelden de genexpressie effectief uit met >50% in de lever en 70% in het jejunum (26).
Een andere benadering voor systemische toediening betreft het gebruik van gespecialiseerde lipidebilayers, stabiele nucleïnezuur-lipide deeltjes (SNALPs) genoemd, waarin chemisch gemodificeerde siRNA’s zijn opgenomen (27). Kationische en neutrale lipiden vormen de bilaag, samen met een buitenste hydrofiele coating van polyethyleenglycol (PEG). In één studie kregen apen een eenmalige dosis van siRNA-bevattende SNALP’s toegediend, die het cholesterolgehalte gedurende 11 dagen of langer verlaagden, waarbij <10% van de APOB-expressie in de lever van dit niet-menselijke primaatmodel overbleef (28). Geen merkbare toxiciteiten werden waargenomen, wat suggereert het potentiële nut van deze methode in systemische delivery.
Voor de in vivo werkzaamheid van siRNA moleculen, de dosering van geleverde siRNAs is een praktische overweging, en selectieve levering van siRNAs aan specifieke weefsels zou mogelijk lager de effectieve dosering nodig. Targeting van receptoren op het celoppervlak is een voordelige aanpak, aangezien hierdoor de siRNA-dosering wordt verlaagd en mogelijk off-target effecten van siRNA-afgifte aan irrelevante weefsels worden vermeden. De koppeling van siRNA’s aan aptameren of antilichaamfragmenten, of het gebruik van nanodeeltjes gecoat met receptorspecifieke liganden, maakt de specifieke toediening van siRNA payloads aan doelcellen en -weefsels mogelijk. Voor het richten van HIV-geïnfecteerde cellen werden siRNA’s gekoppeld aan fragmenten van zware ketenantilichamen (Fabs) die de HIV-omhullende glycoproteïne gp120 herkennen. Positief geladen protamine werd geconjugeerd aan Fab moleculen, en de negatief geladen siRNAs interageerden elektrostatisch met de protamine om een Fab-siRNA complex te vormen. Deze antilichaam-gebaseerde benadering aangetoond >70% knockdown van p24 groep-specifiek antigeen eiwit (Gag) bij het richten gekweekte T lymfocyten geïnfecteerd met HIV-1 (29). Een andere benadering maakte gebruik van een peptide van het hondsdolheidsvirus, dat zich specifiek bindt aan de acetylcholinereceptor. Wanneer dit peptide werd geconjugeerd aan een polyarginine peptide dat siRNA’s bindt, werd levering van siRNA’s aan het centrale zenuwstelsel bereikt, wat resulteerde in remming van een fatale encefalitis virale infectie (30).
Aptameren, die gestructureerde RNA-liganden zijn, kunnen worden ontworpen om specifiek te binden aan receptoren op de celoppervlakte en covalent te worden gekoppeld aan siRNA’s voor specifieke in vivo levering. Bij één methode werd gebruik gemaakt van aptameren die zich binden aan het prostaat-specifiek membraanantigeen (PSMA) dat tot expressie komt op het oppervlak van prostaatkankercellen. Wanneer geconjugeerd aan siRNAs, verminderden deze aptamer-siRNA hybriden effectief de tumorgroei in muizen (31). Een soortgelijke aanpak met behulp van zowel gebiotinyleerde siRNA’s en aptameren gebonden aan het biotine-bindende eiwit streptavidine maakte gebruik van 27-mer siRNA’s om krachtig te induceren gen silencing (32).
Coating nanodeeltjes met celtype-specifieke liganden is een andere krachtige aanpak om systemisch te leveren RNAi-inducerende moleculen. In een belangrijke proof-of-concept studie, werden Ewing sarcoom tumoren gericht in vivo met transferrine ligand-gecoate nanodeeltjes (33). Deze nanodeeltjes werden vervaardigd met behulp van cyclodextrine-bevattende polykationen (CDP’s) die speciaal waren ontworpen om negatief geladen siRNA-moleculen op te nemen. Voor extra stabiliteit en om aggregatie te voorkomen werden PEG polymeren aan het buitenoppervlak bevestigd met behulp van adamantaan eindgroepen. Transferrine-liganden werden vervolgens covalent gekoppeld aan de adamantaan-PEG-ketens, en het nanodeeltjesontwerp maakte zelfassemblage tot uniforme nanodeeltjes van ∼50 nanometer mogelijk. De met nanodeeltjes geïncorporeerde siRNA’s waren gericht tegen het Ews-Fli1 (Ewing sarcoma breakpoint region 1-flightless 1 homolog) genfusieproduct en bleken de vorming van tumor l in muizen te remmen (33).
Conclusie
In samenvatting, de ontwikkeling van de eerste ontdekking van RNAi tot de klinische toepassingen ervan is verbazingwekkend geweest. Inzicht in de fundamentele biologie van RNAi heeft geleid tot wijdverbreide toepassingen in fundamenteel onderzoek en vervolgens tot toepassingen voor de behandeling van ziekten. In de komende jaren zullen naar verwachting meer RNAi-gemedieerde reguleringen van genexpressie worden ontrafeld, en zullen ook op RNAi gebaseerde geneesmiddelen worden goedgekeurd voor gebruik bij de behandeling van ziekten. Bovendien heeft RNAi bewezen een krachtig instrument te zijn voor het bestuderen van de genfunctie en heeft het nieuwe gebieden van fundamenteel onderzoek geopend. In de nabije toekomst zouden we een verdere ontwikkeling moeten zien in ons begrip en de toepassing van dit opmerkelijke cellulaire mechanisme voor posttranscriptionele regulatie van genexpressie.
Acknowledgments
Dit werk werd gesteund door het National Institutes of Health National Institute of Allergy and Infectious Diseases en het NIH Heart Lung and Blood Institute aan J.J.R. D.H.K. wordt ondersteund door een City of Hope pre-doctoral fellowship.
Competing Interests Statement
De auteurs verklaren geen concurrerende belangen.
- 1. Almeida, R. en R.C. Allshire. 2005. RNA silencing and genome regulation. Trends Cell Biol. 15:251-258.Crossref, Medline, CAS, Google Scholar
- 2. Zhang, H., F.A. Kolb, L. Jaskiewicz, E. Westhof, and W. Filipowicz. 2004. Single processing center models for human Dicer and bacterial RNase III. Cell 118:57-68.Crossref, Medline, CAS, Google Scholar
- 3. Lee, Y., I. Hur, S.Y. Park, Y.K. Kim, M.R. Suh, and V.N. Kim. 2006. De rol van PACT in de RNA silencing pathway. EMBO J. 25:522-532.Crossref, Medline, CAS, Google Scholar
- 4. Meister, G., M. Landthaler, A. Patkaniowska, Y. Dorsett, G. Teng, and T. Tuschl. 2004. Human Argonaute2 medieert RNA splitsing gericht door miRNAs en siRNAs. Mol. Cell 15:185-197.Crossref, Medline, CAS, Google Scholar
- 5. Liu, J., M.A. Carmell, F.V. Rivas, C.G. Marsden, J.M. Thomson, J.J. Song, S.M. Hammond, L. Joshua-Tor, et al. 2004. Argonaute2 is de katalytische motor van RNAi bij zoogdieren. Science 305:1437-41.Crossref, Medline, CAS, Google Scholar
- 6. Tang, G. 2005. siRNA and miRNA: an insight into RISCs. Trends Biochem. Sci. 30:106-114.Crossref, Medline, CAS, Google Scholar
- 7. Schwarz, D.S., G. Hutvagner, T. Du, Z. Xu, N. Aronin, and P.D. Zamore. 2003. Asymmetrie in de assemblage van het RNAi-enzymcomplex. Cell 115:199-208.Crossref, Medline, CAS, Google Scholar
- 8. Khvorova, A., A. Reynolds, and S.D. Jayasena. 2003. Functionele siRNAs en miRNAs vertonen strand bias. Cell 115:209-216.Crossref, Medline, CAS, Google Scholar
- 9. Bartel, D.P. 2004. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell 116:281-297.Crossref, Medline, CAS, Google Scholar
- 10. Bartel, D.P. en C.Z. Chen. 2004. Micromanagers of gene expression: the potentially widespread influence of metazoan microRNAs. Nat. Rev. Genet. 5:396-400.Crossref, Medline, CAS, Google Scholar
- 11. Bagga, S., J. Bracht, S. Hunter, K. Massirer, J. Holtz, R. Eachus, and A.E. Pasquinelli. 2005. Regulatie door let-7 en lin-4 miRNAs resulteert in doel-mRNA degradatie. Cell 122:553-563.Crossref, Medline, CAS, Google Scholar
- 12. Leuschner, P.J., S.L. Ameres, S. Kueng, and J. Martinez. 2006. Cleavage of the siRNA passenger strand during RISC assembly in human cells. EMBO Rep. 7:314-320.Crossref, Medline, CAS, Google Scholar
- 13. Gregory, R.I., T.P. Chendrimada, N. Cooch, and R. Shiekhattar. 2005. Human RISC koppelt microRNA biogenese en posttranscriptionele gene silencing. Cell 123:631-640.Crossref, Medline, CAS, Google Scholar
- 14. Matranga, C., Y. Tomari, C. Shin, D.P. Bartel, and P.D. Zamore. 2005. Passenger-strand cleavage vergemakkelijkt assemblage van siRNA in Ago2-bevattende RNAi-enzymcomplexen. Cell 123:607-620.Crossref, Medline, CAS, Google Scholar
- 15. Lee, Y., C. Ahn, J. Han, H. Choi, J. Kim, J. Yim, J. Lee, P. Provost, et al. 2003. Het nucleaire RNase III Drosha initieert microRNA verwerking. Nature 425:415-419.Crossref, Medline, CAS, Google Scholar
- 16. Han, J., Y. Lee, K.H. Yeom, Y.K. Kim, H. Jin, and V.N. Kim. 2004. Het Drosha-DGCR8 complex in primaire microRNA processing. Genes Dev. 18:3016-3027.Crossref, Medline, CAS, Google Scholar
- 17. Hannon, G.J. en J.J. Rossi. 2004. Unlocking the potential of the human genome with RNA interference. Nature 431:371-378.Crossref, Medline, CAS, Google Scholar
- 18. Scherer, L.J. en J.J. Rossi. 2003. Approaches for the sequence-specific knockdown of mRNA. Nat. Biotechnol. 21:1457-1465.Crossref, Medline, CAS, Google Scholar
- 19. Fattal, E. en A. Bochot. 2006. Oculaire levering van nucleïnezuren: antisense oligonucleotiden, aptameren en siRNA. Adv. Drug Deliv. Rev. 58:1203-1223.Crossref, Medline, CAS, Google Scholar
- 20. Tolentino, M. 2006. Interference RNA technology in the treatment of CNV. Ophthalmol. Clin. North Am. 19:393-399 vi-vii.Medline, Google Scholar
- 21. Leonard, J.N. en D.V. Schaffer. 2006. Antiviral RNAi therapy: emerging approaches for hitting a moving target. Gene Ther. 13:532-540.Crossref, Medline, CAS, Google Scholar
- 22. Svoboda, P. 2007. Off-targeting en andere niet-specifieke effecten van RNAi experimenten in zoogdiercellen. Curr. Opin. Mol. Ther. 9:248-257.Medline, CAS, Google Scholar
- 23. Czauderna, F., M. Fechtner, S. Dames, H. Aygun, A. Klippel, G.J. Pronk, K. Giese, and J. Kaufmann. 2003. Structurele variaties en stabiliserende modificaties van synthetische siRNA’s in zoogdiercellen. Nucleic Acids Res. 31:2705-2716.Crossref, Medline, CAS, Google Scholar
- 24. Kim, D.H., M.A. Behlke, S.D. Rose, M.S. Chang, S. Choi, and J.J. Rossi. 2005. Synthetische dsRNA Dicer substraten verhogen RNAi potentie en werkzaamheid. Nat. Biotechnol. 23:222-226.Crossref, Medline, CAS, Google Scholar
- 25. Amarzguioui, M., P. Lundberg, E. Cantin, J. Hagstrom, M.A. Behlke, and J.J. Rossi. 2006. Rationeel ontwerp en in vitro en in vivo levering van Dicer substraat siRNA. Nat. Protocols 1:508-517.Crossref, Medline, CAS, Google Scholar
- 26. Soutschek, J., A. Akinc, B. Bramlage, K. Charisse, R. Constien, M. Donoghue, S. Elbashir, A. Geick, et al. 2004. Therapeutische silencing van een endogeen gen door systemische toediening van gemodificeerde siRNAs. Nature 432:173-178.Crossref, Medline, CAS, Google Scholar
- 27. Morrissey, D.V., J.A. Lockridge, L. Shaw, K. Blanchard, K. Jensen, W. Breen, K. Hartsough, L. Machemer, et al. 2005. Potente en persistente in vivo anti-HBV activiteit van chemisch gemodificeerde siRNAs. Nat. Biotechnol. 23:1002-1007.Crossref, Medline, CAS, Google Scholar
- 28. Zimmermann, T.S., A.C. Lee, A. Akinc, B. Bramlage, D. Bumcrot, M.N. Fedoruk, J. Harborth, J.A. Heyes, et al. 2006. RNAi-gemedieerde gene silencing bij niet-menselijke primaten. Nature 441:111-114.Crossref, Medline, CAS, Google Scholar
- 29. Song, E., P. Zhu, S.K. Lee, D. Chowdhury, S. Kussman, D.M. Dykxhoorn, Y. Feng, D. Palliser, et al. 2005. Antilichaam gemedieerde in vivo levering van kleine interfererende RNA’s via cel-oppervlakte receptoren. Nat. Biotechnol. 23:709-717.Crossref, Medline, CAS, Google Scholar
- 30. Kumar, P., H. Wu, J.L. McBride, K.E. Jung, M.H. Kim, B.L. Davidson, S.K. Lee, P. Shankar, et al. 2007. Transvasculaire levering van klein interfererend RNA aan het centrale zenuwstelsel. Nature 448:39-43.Crossref, Medline, CAS, Google Scholar
- 31. McNamara, J.O., II, E.R. Andrechek, Y. Wang, K.D. Viles, R.E. Rempel, E. Gilboa, B.A. Sullenger, and P.H. Giangrande. 2006. Celtype-specifieke levering van siRNAs met aptamer-siRNA chimeras. Nat. Biotechnol. 24:1005-1015.Crossref, Medline, CAS, Google Scholar
- 32. Chu, T.C., K.Y. Twu, A.D. Ellington, and M. Levy. 2006. Aptamer gemedieerde siRNA levering. Nucleic Acids Res. 34:e73.Crossref, Medline, Google Scholar
- 33. Hu-Lieskovan, S., J.D. Heidel, D.W. Bartlett, M.E. Davis, and T.J. Triche. 2005. Sequence-specific knockdown of EWS-FLI1 by targeted, nonviral delivery of small interfering RNA inhibits tumor growth in a murine model of metastatic Ewing’s sarcoma. Cancer Res. 65:8984-8992.Crossref, Medline, CAS, Google Scholar