Introducción
El ARN de interferencia (ARNi) es un mecanismo regulador de la mayoría de las células eucariotas que utiliza pequeñas moléculas de ARN de doble cadena (ARNd) como desencadenantes del control directo dependiente de la homología de la actividad génica (Figura 1) (1). Conocidas como pequeños ARN de interferencia (ARNsi), estas moléculas de ARNd de ∼21-22 pb de longitud tienen unos voladizos 3′ característicos de 2 nt que les permiten ser reconocidos por la maquinaria enzimática del ARNi, lo que finalmente conduce a la degradación dependiente de la homología del ARNm objetivo. En las células de mamíferos, los siRNAs se producen a partir de la escisión de precursores de dsRNA más largos por la endonucleasa RNasa III Dicer (2), o pueden sintetizarse por métodos químicos o bioquímicos. Dicer forma un complejo con las proteínas de unión al ARN, la proteína de unión al ARN-TAR (TRBP), PACT y Ago-2, que participan en la transferencia de los ARNsi al complejo de silenciamiento inducido por ARN (RISC) (3). Los componentes principales del RISC son los miembros de la familia Argonauta (Ago). En los humanos hay ocho miembros de esta familia, pero sólo Ago-2 posee un dominio catalítico activo para la actividad de corte (4,5). Mientras que los siRNAs cargados en RISC son de doble cadena, Ago-2 escinde y libera la cadena «pasajera», dando lugar a una forma activada de RISC con una molécula de ARN «guía» de una sola cadena que dirige la especificidad del reconocimiento de la diana mediante el emparejamiento de bases intermolecular (6). Las reglas que rigen la selectividad de la carga de la hebra en el RISC se basan en las estabilidades termodinámicas diferenciales de los extremos de los siRNAs (7,8). El extremo menos estable termodinámicamente se ve favorecido para unirse al dominio PIWI de Ago-2.
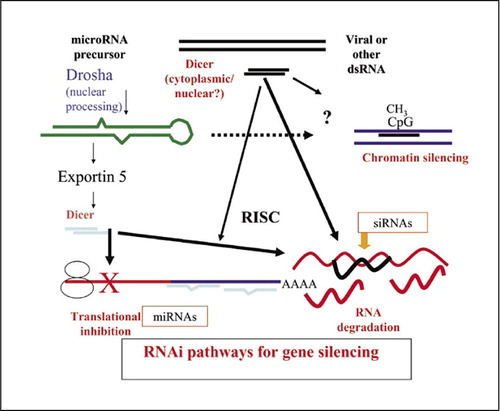
El ARNi es multifacético, y existen varias vías en las que pequeños ARN de doble cadena (ARNd) regulan la expresión génica. La vía del microARN endógeno (miARN) comienza con los miARN primarios transcritos por Pol II que se procesan en el núcleo hasta convertirse en pre-miARN, se exportan al citoplasma y se procesan de nuevo hasta convertirse en miARN funcionales. La función principal de los miARNs es inhibir la traducción mediante el emparejamiento incompleto de bases Watson-Crick con las regiones no traducidas 3′ de los ARNm objetivo. Como alternativa, se pueden producir pequeños ARN de interferencia (siRNA) perfectamente duplexados de forma intracelular o suministrados de forma exógena a las células. La cadena guía se incorpora al complejo de silenciamiento inducido por ARN (RISC), donde guía la degradación específica de la secuencia del transcrito objetivo, independientemente de dónde se produzca el emparejamiento de bases. Las vías del miARN y del siARN son intercambiables, y los determinantes importantes son las posiciones dentro del mensaje y el grado de emparejamiento de bases con los transcritos objetivo. Los siRNAs también pueden desencadenar el silenciamiento transcripcional de los genes a través de interacciones con la cromatina, en las que guían la metilación de las histonas y del ADN, lo que conduce a una cromatina inactiva.
MicroRNAs
Una rama importante del RNAi implica a los microRNAs (miRNAs). Se trata de dúplex endógenos que regulan post-transcripcionalmente la expresión génica mediante la formación de complejos con RISC y la unión a las regiones 3′ no traducidas (UTRs) de las secuencias diana a través de tramos cortos de homología, denominados «secuencias semilla» (9,10). El principal mecanismo de acción de los miRNAs es la represión traslacional, aunque ésta puede ir acompañada de la degradación del mensaje (11). Los dúplex de miARN poseen un emparejamiento de bases Watson-Crick incompleto, y la cadena antisentido no puede elegirse por escisión de la cadena pasajera como ocurre con los siARN; por tanto, la cadena antisentido debe elegirse por un mecanismo alternativo (12-14). Los miARN son sustratos endógenos para la maquinaria del ARNi. Se expresan inicialmente como transcritos primarios largos (pri-miRNAs), que son procesados dentro del núcleo en horquillas de 60-70 pb por el complejo Microprocesador, formado por Drosha y DGCR8 (15,16) en pre-miRNAs. Los pre-miRNAs son procesados posteriormente en el citoplasma por Dicer y una de las dos hebras se carga en el RISC, presumiblemente a través de la interacción con una de las proteínas accesorias de Dicer (3). Es importante destacar que es posible explotar esta vía nativa de silenciamiento génico para la regulación del gen o genes que se desee. Si el efector de ARNsi se introduce en la célula, «activará» el RISC, lo que dará lugar a un silenciamiento potente y específico del ARNm objetivo. Debido a la potencia y selectividad del ARNi, se ha convertido en la metodología de elección para silenciar la expresión de genes específicos en las células de mamíferos.
El ARNi como enfoque terapéutico para el tratamiento de enfermedades
El control de los genes asociados a las enfermedades hace que el ARNi sea una opción atractiva para futuras terapias. Básicamente, todas las enfermedades humanas causadas por la actividad de uno o unos pocos genes deberían ser susceptibles de una intervención basada en el ARNi. Esta lista incluye el cáncer, las enfermedades autoinmunes, los trastornos genéticos dominantes y las infecciones virales. El ARNi puede activarse por dos vías diferentes: (i) un enfoque basado en el ARN en el que los ARNsi efectores sintéticos son entregados por varios portadores a las células diana como dúplex preformados de 21 bases; o (ii) a través de estrategias basadas en el ADN en las que los efectores de ARNsi son producidos por el procesamiento intracelular de transcripciones de horquillas de ARN más largas (revisado en las referencias 17 y 18). Este último enfoque se basa principalmente en la síntesis nuclear de ARN de horquilla corta (ARNhc), que son transportados al citoplasma a través de la vía de exportación de miARN y son procesados en ARNsi por Dicer. Aunque el uso directo de efectores de ARNsi sintéticos es sencillo y suele dar lugar a un potente silenciamiento génico, el efecto es transitorio. Los fármacos de ARNi basados en el ADN, en cambio, tienen el potencial de introducirse de forma estable cuando se utilizan en un entorno de terapia génica, lo que permite, en principio, un único tratamiento de genes de ARNhc administrados por vectores virales.
Las primeras aplicaciones clínicas del ARNi se han dirigido al tratamiento de la degeneración macular asociada a la edad (DMAE), que causa ceguera o visión limitada en millones de adultos anualmente (19,20). También se están desarrollando terapias basadas en el ARNi para las infecciones víricas, como el virus de la inmunodeficiencia humana (VIH), los virus de la hepatitis B y C (VHB y VHC) y el virus respiratorio sincitial (VRS) (21). También están en marcha estrategias para el tratamiento de enfermedades neurodegenerativas y cánceres.
Aunque los estudios in vivo realizados con éxito han demostrado la eficacia potencial de las terapias basadas en el ARNi, otros estudios han ilustrado enfoques específicos que deben evitarse al adoptar un mecanismo celular endógeno para obtener beneficios terapéuticos. Los efectos secundarios no deseados han incluido la activación de los receptores tipo Toll (TLR) y las respuestas de interferón tipo 1, y la competencia con los componentes de la vía endógena del ARNi (22). Estos hallazgos indican que, aunque el ARNi es un mecanismo potencialmente revolucionario para el tratamiento de enfermedades, es necesario tener la debida precaución al interpretar los resultados de las eliminaciones de dianas mediadas por ARNi.
El reto de la administración de ARNi específica para cada célula o tejido también es crucial a la hora de investigar la utilidad de las terapias basadas en ARNi para una enfermedad determinada; varias estrategias para la administración no viral y viral de los activadores de ARNi han demostrado ser eficaces en sus respectivos modelos de enfermedad. Las ventajas y desventajas relativas del uso de siRNAs sintéticos frente a los shRNAs expresados también deben tenerse en cuenta a la hora de diseñar terapias basadas en el RNAi para una enfermedad concreta.
Los siRNAs sintetizados químicamente se examinan habitualmente para conseguir el derribo efectivo de un gen diana específico. Para aumentar la estabilidad del siRNA, se introducen modificaciones químicas, como las 2′-O-metilpurinas o las 2′-fluoropirimidinas (23). Cuando se diseña inicialmente una molécula de ARNsi, se utilizan rutinariamente algoritmos computacionales que incorporan varios parámetros, incluyendo las estabilidades de los extremos del dúplex de ARNsi para la selección adecuada de la cadena y las estructuras secundarias del ARNm para la accesibilidad al sitio objetivo. Para mejorar la potencia de una respuesta de ARNi, los dúplex de ARNsi también pueden diseñarse para imitar sustratos para el procesamiento de Dicer (24). Los siRNAs más largos (por ejemplo, de 27 mers) se incorporan en el paso de carga de Dicer de la vía del RNAi y pueden facilitar la activación de RISC. Los 27-mers están diseñados asimétricamente para mostrar un voladizo de 2 nt 3′ en un extremo y una región roma en el otro (25), lo que guía el procesamiento de Dicer y la biogénesis de la cadena guía adecuada, ya que el dominio PAZ de Dicer reconoce el extremo del voladizo. Además, debido a la eficiencia de los 27-mers en la mediación del silenciamiento génico, una menor concentración de siRNAs puede mediar una potente respuesta de RNAi.
Desde la perspectiva de las aplicaciones terapéuticas de RNAi, la preocupación más importante es la entrega de los siRNAs al tejido apropiado. Numerosas publicaciones recientes han demostrado que los siRNAs pueden ser administrados sistémicamente en varios tejidos con el consiguiente derribo de los RNAs objetivo. La inyección intravenosa de ARNsi para su administración sistémica se realiza mediante la conjugación de moléculas de ARNsi con un grupo de colesterol o el empaquetamiento de ARNsi en partículas liposomales. La administración sistémica mediante estos métodos es eficaz para la administración en el hígado y el yeyuno, pero puede no ser apropiada para la administración en otros órganos. En un estudio de prueba de concepto, se utilizaron ARNsi dirigidos a la apolipoproteína B (APOB) para modificar el metabolismo del colesterol. El grupo hidroxilo 3′ de la hebra pasajera del siRNA se unió químicamente a un grupo de colesterol, y estos siRNAs conjugados redujeron eficazmente la expresión génica en un >50% en el hígado y en un 70% en el yeyuno (26).
Otro enfoque para la administración sistémica implica el uso de bicapas lipídicas especializadas denominadas partículas estables de ácido nucleico-lípido (SNALPs), que incorporan siRNAs químicamente modificados (27). Los lípidos catiónicos y neutros componen la bicapa, junto con un revestimiento exterior hidrofílico de polietilenglicol (PEG). En un estudio, se administró a monos una dosis única de SNALP con ARNsi, que redujo los niveles de colesterol durante 11 días o más, con un <10% de expresión de APOB que permaneció en el hígado de este modelo de primate no humano (28). No se observaron toxicidades notables, lo que sugiere la utilidad potencial de este método en la administración sistémica.
Para la eficacia in vivo de las moléculas de ARNsi, la dosis de ARNsi administrada es una consideración práctica, y la administración selectiva de ARNsi a tejidos específicos podría reducir la dosis efectiva requerida. Dirigirse a los receptores de la superficie celular es un enfoque ventajoso, ya que reduciría la dosis de siRNA y evitaría potencialmente los efectos fuera del objetivo de la entrega de siRNA a tejidos irrelevantes. El acoplamiento de ARNsi a aptámeros o fragmentos de anticuerpos, o el uso de nanopartículas recubiertas con ligandos específicos de receptores, permite la entrega específica de cargas útiles de ARNsi a las células y tejidos objetivo. Para dirigirse a las células infectadas por el VIH, los siRNAs se acoplaron a fragmentos de anticuerpos de cadena pesada (Fabs) que reconocen la glicoproteína de envoltura del VIH gp120. La protamina cargada positivamente se conjugó con las moléculas Fab, y los siRNAs cargados negativamente interactuaron electrostáticamente con la protamina para formar un complejo Fab-siRNA. Este enfoque basado en anticuerpos demostró un >70% de eliminación de la proteína del antígeno específico del grupo p24 (Gag) cuando se dirigió a linfocitos T cultivados infectados con el VIH-1 (29). Un enfoque de orientación diferente aprovechó un péptido del virus de la rabia, que se une específicamente al receptor de la acetilcolina. Cuando este péptido se conjugó con un péptido de poliarginina que se une a los siRNAs, se logró la entrega de siRNAs al sistema nervioso central, lo que resultó en la inhibición de una infección viral de encefalitis fatal (30).
Los aptámeros, que son ligandos de ARN estructurados, pueden diseñarse para unirse específicamente a los receptores de la superficie celular y unirse covalentemente a los siRNAs para la entrega específica in vivo. Un método utilizó aptámeros que se unen al antígeno de membrana específico de la próstata (PSMA) expresado en la superficie de las células cancerosas de la próstata. Cuando se conjugan con ARNsi, estos híbridos aptámero-ARNsi reducen eficazmente el crecimiento del tumor en ratones (31). Un enfoque similar, en el que se utilizaron tanto siRNAs biotinilados como aptámeros unidos a la proteína de unión a biotina estreptavidina, permitió utilizar siRNAs de 27 marcadores para inducir de forma potente el silenciamiento de genes (32).
El recubrimiento de nanopartículas con ligandos específicos para cada tipo de célula es otro potente enfoque para la administración sistémica de moléculas inductoras de RNAi. En un importante estudio de prueba de concepto, los tumores de sarcoma de Ewing fueron atacados in vivo con nanopartículas recubiertas de ligando de transferrina (33). Estas nanopartículas se construyeron utilizando policationes que contenían ciclodextrina (CDP) diseñadas específicamente para incorporar moléculas de ARNsi con carga negativa. Para aumentar la estabilidad y evitar la agregación, se unieron polímeros de PEG a la superficie exterior mediante grupos terminales de adamantano. A continuación, los ligandos de transferrina se unieron covalentemente a las cadenas de adamantano y PEG, y el diseño de las nanopartículas permitió su autoensamblaje en nanopartículas uniformes de tamaño ∼50 nanómetros. Los ARNsi incorporados a las nanopartículas se dirigían al producto de fusión génica Ews-Fli1 (Ewing sarcoma breakpoint region 1-flightless 1 homolog) y demostraron inhibir la formación de tumores en ratones (33).
Observaciones finales
En resumen, la progresión desde el descubrimiento inicial del ARNi hasta sus aplicaciones clínicas ha sido asombrosa. La comprensión de la biología fundamental del ARNi ha conducido a sus aplicaciones generalizadas en la investigación básica y, posteriormente, en las aplicaciones para el tratamiento de enfermedades. En los próximos años cabe esperar que se desvele más regulación de la expresión génica mediada por el ARNi, y también que se aprueben fármacos basados en el ARNi para su uso en el tratamiento de enfermedades. Además, el ARNi ha demostrado ser una poderosa herramienta para el estudio de la función génica y ha abierto nuevas áreas de investigación básica. En un futuro próximo deberíamos ver un desarrollo continuo en nuestra comprensión y aplicación de este notable mecanismo celular para la regulación postranscripcional de la expresión génica.
Agradecimientos
Este trabajo fue apoyado por los Institutos Nacionales de Salud Instituto Nacional de Alergias y Enfermedades Infecciosas y el Instituto del Corazón, Pulmón y Sangre de los NIH a J.J.R. D.H.K. cuenta con el apoyo de una beca predoctoral de City of Hope.
Declaración de intereses en competencia
Los autores declaran no tener intereses en competencia.
- 1. Almeida, R. y R.C. Allshire. 2005. Silenciamiento del ARN y regulación del genoma. Trends Cell Biol. 15:251-258.Crossref, Medline, CAS, Google Scholar
- 2. Zhang, H., F.A. Kolb, L. Jaskiewicz, E. Westhof, y W. Filipowicz. 2004. Single processing center models for human Dicer and bacterial RNase III. Cell 118:57-68.Crossref, Medline, CAS, Google Scholar
- 3. Lee, Y., I. Hur, S.Y. Park, Y.K. Kim, M.R. Suh, y V.N. Kim. 2006. The role of PACT in the RNA silencing pathway. EMBO J. 25:522-532.Crossref, Medline, CAS, Google Scholar
- 4. Meister, G., M. Landthaler, A. Patkaniowska, Y. Dorsett, G. Teng, y T. Tuschl. 2004. Human Argonaute2 mediates RNA cleavage targeted by miRNAs and siRNAs. Mol. Cell 15:185-197.Crossref, Medline, CAS, Google Scholar
- 5. Liu, J., M.A. Carmell, F.V. Rivas, C.G. Marsden, J.M. Thomson, J.J. Song, S.M. Hammond, L. Joshua-Tor, et al. 2004. Argonaute2 es el motor catalítico del RNAi en mamíferos. Science 305:1437-41.Crossref, Medline, CAS, Google Scholar
- 6. Tang, G. 2005. siRNA and miRNA: an insight into RISCs. Trends Biochem. Sci. 30:106-114.Crossref, Medline, CAS, Google Scholar
- 7. Schwarz, D.S., G. Hutvagner, T. Du, Z. Xu, N. Aronin, and P.D. Zamore. 2003. Asimetría en el ensamblaje del complejo enzimático del ARNi. Cell 115:199-208.Crossref, Medline, CAS, Google Scholar
- 8. Khvorova, A., A. Reynolds, y S.D. Jayasena. 2003. Functional siRNAs and miRNAs exhibit strand bias. Cell 115:209-216.Crossref, Medline, CAS, Google Scholar
- 9. Bartel, D.P. 2004. MicroRNAs: genómica, biogénesis, mecanismo y función. Cell 116:281-297.Crossref, Medline, CAS, Google Scholar
- 10. Bartel, D.P. y C.Z. Chen. 2004. Micromanagers of gene expression: the potentially widespread influence of metazoan microRNAs. Nat. Rev. Genet. 5:396-400.Crossref, Medline, CAS, Google Scholar
- 11. Bagga, S., J. Bracht, S. Hunter, K. Massirer, J. Holtz, R. Eachus y A.E. Pasquinelli. 2005. Regulation by let-7 and lin-4 miRNAs results in target mRNA degradation. Cell 122:553-563.Crossref, Medline, CAS, Google Scholar
- 12. Leuschner, P.J., S.L. Ameres, S. Kueng, y J. Martinez. 2006. Cleavage of the siRNA passenger strand during RISC assembly in human cells. EMBO Rep. 7:314-320.Crossref, Medline, CAS, Google Scholar
- 13. Gregory, R.I., T.P. Chendrimada, N. Cooch, y R. Shiekhattar. 2005. El RISC humano acopla la biogénesis de microARN y el silenciamiento génico postranscripcional. Cell 123:631-640.Crossref, Medline, CAS, Google Scholar
- 14. Matranga, C., Y. Tomari, C. Shin, D.P. Bartel y P.D. Zamore. 2005. Passenger-strand cleavage facilitates assembly of siRNA into Ago2-containing RNAi enzyme complexes. Cell 123:607-620.Crossref, Medline, CAS, Google Scholar
- 15. Lee, Y., C. Ahn, J. Han, H. Choi, J. Kim, J. Yim, J. Lee, P. Provost, et al. 2003. The nuclear RNase III Drosha initiates microRNA processing. Nature 425:415-419.Crossref, Medline, CAS, Google Scholar
- 16. Han, J., Y. Lee, K.H. Yeom, Y.K. Kim, H. Jin y V.N. Kim. 2004. El complejo Drosha-DGCR8 en el procesamiento primario de microARN. Genes Dev. 18:3016-3027.Crossref, Medline, CAS, Google Scholar
- 17. Hannon, G.J. y J.J. Rossi. 2004. Desbloqueando el potencial del genoma humano con la interferencia del ARN. Nature 431:371-378.Crossref, Medline, CAS, Google Scholar
- 18. Scherer, L.J. y J.J. Rossi. 2003. Approaches for the sequence-specific knockdown of mRNA. Nat. Biotechnol. 21:1457-1465.Crossref, Medline, CAS, Google Scholar
- 19. Fattal, E. y A. Bochot. 2006. Ocular delivery of nucleic acids: antisense oligonucleotides, aptamers and siRNA. Adv. Drug Deliv. Rev. 58:1203-1223.Crossref, Medline, CAS, Google Scholar
- 20. Tolentino, M. 2006. Tecnología de ARN de interferencia en el tratamiento de la NVC. Ophthalmol. Clin. North Am. 19:393-399 vi-vii.Medline, Google Scholar
- 21. Leonard, J.N. y D.V. Schaffer. 2006. Antiviral RNAi therapy: emerging approaches for hitting a moving target. Gene Ther. 13:532-540.Crossref, Medline, CAS, Google Scholar
- 22. Svoboda, P. 2007. Off-targeting and other non-specific effects of RNAi experiments in mammalian cells. Curr. Opin. Mol. Ther. 9:248-257.Medline, CAS, Google Scholar
- 23. Czauderna, F., M. Fechtner, S. Dames, H. Aygun, A. Klippel, G.J. Pronk, K. Giese y J. Kaufmann. 2003. Structural variations and stabilising modifications of synthetic siRNAs in mammalian cells. Nucleic Acids Res. 31:2705-2716.Crossref, Medline, CAS, Google Scholar
- 24. Kim, D.H., M.A. Behlke, S.D. Rose, M.S. Chang, S. Choi y J.J. Rossi. 2005. Synthetic dsRNA Dicer substrates enhance RNAi potency and efficacy. Nat. Biotechnol. 23:222-226.Crossref, Medline, CAS, Google Scholar
- 25. Amarzguioui, M., P. Lundberg, E. Cantin, J. Hagstrom, M.A. Behlke y J.J. Rossi. 2006. Rational design and in vitro and in vivo delivery of Dicer substrate siRNA. Nat. Protocols 1:508-517.Crossref, Medline, CAS, Google Scholar
- 26. Soutschek, J., A. Akinc, B. Bramlage, K. Charisse, R. Constien, M. Donoghue, S. Elbashir, A. Geick, et al. 2004. Silenciamiento terapéutico de un gen endógeno mediante la administración sistémica de siRNAs modificados. Nature 432:173-178.Crossref, Medline, CAS, Google Scholar
- 27. Morrissey, D.V., J.A. Lockridge, L. Shaw, K. Blanchard, K. Jensen, W. Breen, K. Hartsough, L. Machemer, et al. 2005. Potente y persistente actividad in vivo contra el VHB de siRNAs químicamente modificados. Nat. Biotechnol. 23:1002-1007.Crossref, Medline, CAS, Google Scholar
- 28. Zimmermann, T.S., A.C. Lee, A. Akinc, B. Bramlage, D. Bumcrot, M.N. Fedoruk, J. Harborth, J.A. Heyes, et al. 2006. RNAi-mediated gene silencing in non-human primates. Nature 441:111-114.Crossref, Medline, CAS, Google Scholar
- 29. Song, E., P. Zhu, S.K. Lee, D. Chowdhury, S. Kussman, D.M. Dykxhoorn, Y. Feng, D. Palliser, et al. 2005. Antibody mediated in vivo delivery of small interfering RNAs via cell-surface receptors. Nat. Biotechnol. 23:709-717.Crossref, Medline, CAS, Google Scholar
- 30. Kumar, P., H. Wu, J.L. McBride, K.E. Jung, M.H. Kim, B.L. Davidson, S.K. Lee, P. Shankar, et al. 2007. Transvascular delivery of small interfering RNA to the central nervous system. Nature 448:39-43.Crossref, Medline, CAS, Google Scholar
- 31. McNamara, J.O., II, E.R. Andrechek, Y. Wang, K.D. Viles, R.E. Rempel, E. Gilboa, B.A. Sullenger y P.H. Giangrande. 2006. Cell type-specific delivery of siRNAs with aptamer-siRNA chimeras. Nat. Biotechnol. 24:1005-1015.Crossref, Medline, CAS, Google Scholar
- 32. Chu, T.C., K.Y. Twu, A.D. Ellington, y M. Levy. 2006. Entrega de siRNA mediada por aptámeros. Nucleic Acids Res. 34:e73.Crossref, Medline, Google Scholar
- 33. Hu-Lieskovan, S., J.D. Heidel, D.W. Bartlett, M.E. Davis y T.J. Triche. 2005. Sequence-specific knockdown of EWS-FLI1 by targeted, nonviral delivery of small interfering RNA inhibits tumor growth in a murine model of metastatic Ewing sarcoma. Cancer Res. 65:8984-8992.Crossref, Medline, CAS, Google Scholar