Wprowadzenie
Interferencja RNA (RNAi) jest mechanizmem regulacyjnym większości komórek eukariotycznych, który wykorzystuje małe dwuniciowe cząsteczki RNA (dsRNA) jako wyzwalacze do bezpośredniej, zależnej od homologii kontroli aktywności genów (Rysunek 1) (1). Znane jako małe interferujące RNA (siRNA), te cząsteczki dsRNA o długości ∼21-22 bp mają charakterystyczne 2 nt 3′ nawisy, które umożliwiają ich rozpoznanie przez enzymatyczną maszynerię RNAi, co ostatecznie prowadzi do zależnej od homologii degradacji docelowego mRNA. W komórkach ssaków siRNA powstają w wyniku rozszczepienia dłuższych prekursorów dsRNA przez endonukleazę RNase III Dicer (2) lub mogą być syntetyzowane metodami chemicznymi lub biochemicznymi. Dicer łączy się z białkami wiążącymi RNA, TAR-RNA-binding protein (TRBP), PACT i Ago-2, które biorą udział w przekazywaniu siRNA do kompleksu wyciszania indukowanego RNA (RISC) (3). Głównymi składnikami RISC są członkowie rodziny Argonaute (Ago). U ludzi istnieje osiem członków tej rodziny, ale tylko Ago-2 posiada aktywną domenę katalityczną dla aktywności rozszczepiającej (4,5). Podczas gdy siRNA ładowane do RISC są dwuniciowe, Ago-2 rozszczepia i uwalnia nić „pasażerską”, co prowadzi do powstania aktywowanej formy RISC z jednoniciową cząsteczką „przewodniego” RNA, która kieruje specyficznością rozpoznawania celu przez międzycząsteczkowe parowanie zasad (6). Zasady rządzące selektywnością ładowania nici do RISC oparte są na różnej termodynamicznej stabilności końców siRNA (7,8). Mniej stabilny termodynamicznie koniec jest preferowany do wiązania z domeną PIWI Ago-2.
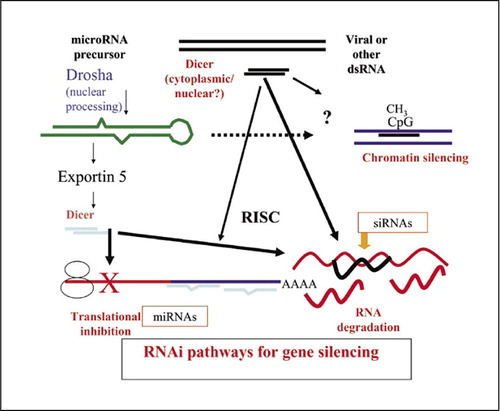
RNAi jest wielopłaszczyznowe i istnieją różne szlaki, w których małe dwuniciowe RNA (dsRNA) regulują ekspresję genów. Szlak endogennego mikro-RNA (miRNA) rozpoczyna się od transkrybowanych przez Pol II pierwotnych miRNA, które są przetwarzane w jądrze do pre-miRNA, eksportowane do cytoplazmy i ponownie przetwarzane w funkcjonalne miRNA. Podstawową funkcją miRNA jest hamowanie translacji poprzez niekompletne parowanie zasad Watsona-Cricka z 3′ nieulegającymi translacji regionami docelowych mRNA. Alternatywnie, doskonale zduplikowane małe interferujące RNA (siRNA) mogą być produkowane wewnątrzkomórkowo lub dostarczane egzogennie do komórek. Nić prowadząca jest włączana do kompleksu wyciszającego indukowanego przez RNA (RISC), gdzie kieruje specyficzną sekwencyjnie degradacją docelowego transkryptu, niezależnie od miejsca parowania zasad. Szlaki miRNA i siRNA są wymienne, a ważnymi wyznacznikami są pozycje w przekazie i stopień sparowania zasad z docelowymi transkryptami. siRNA mogą również wyzwalać transkrypcyjne wyciszanie genów poprzez interakcje z chromatyną, gdzie kierują metylacją histonów i DNA prowadząc do nieaktywnej chromatyny.
MikroRNA
Ważne ramię RNAi obejmuje mikroRNA (miRNA). Są to endogenne dupleksy, które potranskrypcyjnie regulują ekspresję genów poprzez łączenie się z RISC i wiązanie się z 3′ regionami nieulegającymi translacji (UTR) sekwencji docelowych za pomocą krótkich odcinków homologicznych, zwanych „sekwencjami zalążkowymi” (9,10). Podstawowym mechanizmem działania miRNA jest represja translacji, choć może jej towarzyszyć degradacja wiadomości (11). Dupleksy miRNA posiadają niekompletne parowanie zasad Watsona-Cricka, a nić antysensowna nie może być wybrana poprzez rozszczepienie nici pasażerskiej, jak to ma miejsce w przypadku siRNA; dlatego nić antysensowna musi być wybrana za pomocą alternatywnego mechanizmu (12-14). miRNA są endogennymi substratami dla maszynerii RNAi. Początkowo ulegają ekspresji jako długie transkrypty pierwotne (pri-miRNA), które w jądrze są przetwarzane przez kompleks mikroprocesorowy, składający się z Drosha i DGCR8 (15,16), na premiRNA o długości 60-70 bp na szpilki do włosów. PremiRNA są dalej przetwarzane w cytoplazmie przez Dicer, a jedna z dwóch nici jest ładowana do RISC, prawdopodobnie poprzez interakcję z jednym z białek pomocniczych Dicer (3). Co ważne, możliwe jest wykorzystanie tego natywnego szlaku wyciszania genów do regulacji wybranego genu (genów). Jeśli efektor siRNA zostanie dostarczony do komórki, „aktywuje” RISC, powodując silne i specyficzne wyciszenie docelowego mRNA. Ze względu na siłę działania i selektywność RNAi, stało się ono metodologią z wyboru do wyciszania specyficznej ekspresji genów w komórkach ssaków.
RNAi jako podejście terapeutyczne do leczenia chorób
Kontrola genów związanych z chorobami czyni RNAi atrakcyjnym wyborem dla przyszłych terapii. W zasadzie każda choroba człowieka spowodowana aktywnością jednego lub kilku genów powinna być podatna na interwencję opartą na RNAi. Lista ta obejmuje nowotwory, choroby autoimmunologiczne, dominujące zaburzenia genetyczne i infekcje wirusowe. RNAi może być wywoływane przez dwie różne ścieżki: (i) podejście oparte na RNA, w którym syntetyczne efektorowe siRNA są dostarczane przez różne nośniki do komórek docelowych jako wstępnie uformowane dupleksy 21 zasad; lub (ii) poprzez strategie oparte na DNA, w których efektory siRNA są wytwarzane przez wewnątrzkomórkowe przetwarzanie dłuższych transkryptów RNA typu szpilka do włosów (omówione w piśmiennictwie 17 i 18). To ostatnie podejście opiera się głównie na jądrowej syntezie RNA o krótkiej szpilce (shRNA), które są transportowane do cytoplazmy przez szlak eksportu miRNA i przetwarzane na siRNA przez Dicer. Bezpośrednie zastosowanie syntetycznych efektorów siRNA jest proste i zwykle prowadzi do silnego wyciszenia genów, jednak efekt jest przejściowy. Z drugiej strony, leki RNAi oparte na DNA mogą być wprowadzane w sposób stabilny, gdy są stosowane w terapii genowej, co pozwala w zasadzie na jednorazowe leczenie genów shRNA dostarczonych przez wektor wirusowy.
Pierwsze kliniczne zastosowania RNAi zostały ukierunkowane na leczenie zwyrodnienia plamki żółtej związanego z wiekiem (AMD), które powoduje ślepotę lub ograniczenie widzenia u milionów dorosłych rocznie (19,20). Obecnie opracowywane są również terapie oparte na RNAi w leczeniu infekcji wirusowych, w tym ludzkiego wirusa niedoboru odporności (HIV), wirusów zapalenia wątroby typu B i C (HBV i HCV) oraz wirusa syncytialnego układu oddechowego (RSV) (21). Trwają również prace nad strategiami leczenia chorób neurodegeneracyjnych i nowotworów.
Pomimo że udane badania in vivo wykazały potencjalną skuteczność terapii opartych na RNAi, inne badania zilustrowały specyficzne podejścia, których należy unikać przy przyjmowaniu endogennego mechanizmu komórkowego dla korzyści terapeutycznych. Niepożądane efekty uboczne obejmowały aktywację receptorów Toll-podobnych (TLRs) i odpowiedzi interferonu typu 1 oraz konkurencję z endogennymi komponentami szlaku RNAi (22). Wyniki te wskazują, że chociaż RNAi jest potencjalnie rewolucyjnym mechanizmem leczenia chorób, konieczna jest należyta ostrożność przy interpretowaniu wyników knockdownu z użyciem RNAi.
Wyzwanie dostarczenia siRNA specyficznego dla komórki lub tkanki jest również kluczowe przy badaniu użyteczności terapii opartych na RNAi w danej chorobie; różne strategie niewirusowego i wirusowego dostarczania wyzwalaczy RNAi okazały się skuteczne w odpowiednich modelach chorób. Względne zalety i wady stosowania syntetycznych siRNA w porównaniu z wyrażonymi shRNA muszą być również brane pod uwagę przy projektowaniu terapii opartych na RNAi dla danej choroby.
Syntetyzowane chemicznie siRNA są powszechnie badane pod kątem efektywnego znokautowania określonego genu docelowego. Aby zwiększyć stabilność siRNA, wprowadza się modyfikacje chemiczne, takie jak 2′-O-metylopuryny lub 2′-fluoropirymidyny (23). Podczas wstępnego projektowania cząsteczki siRNA rutynowo stosuje się algorytmy obliczeniowe, które uwzględniają różne parametry, w tym stabilność końców dupleksu siRNA w celu właściwego doboru nici oraz struktury drugorzędowe mRNA w celu zapewnienia dostępności miejsca docelowego. Aby poprawić siłę reakcji RNAi, dupleksy siRNA mogą być również zaprojektowane tak, aby naśladować substraty do przetwarzania przez Dicer (24). Dłuższe siRNA (np. 27-merowe) są włączane do etapu ładowania Dicer szlaku RNAi i mogą ułatwiać aktywację RISC. 27-mery są zaprojektowane asymetrycznie, aby wykazywać 2 nt 3′ nawisu na jednym końcu i tępy region na drugim (25), co kieruje przetwarzaniem Dicer i biogenezą właściwej nici prowadzącej, ponieważ domena PAZ Dicer rozpoznaje koniec nawisu. Ponadto, ze względu na wydajność 27-merów w wyciszaniu genów, niższe stężenie siRNA może pośredniczyć w silnej odpowiedzi RNAi.
Z punktu widzenia terapeutycznych zastosowań RNAi, najważniejszym problemem jest dostarczenie siRNA do odpowiedniej tkanki. Liczne ostatnie publikacje wykazały, że siRNA mogą być systemowo dostarczane do różnych tkanek, co skutkuje obniżeniem docelowego RNA. Dożylne wstrzykiwanie siRNA w celu systemowego dostarczania jest realizowane poprzez koniugację cząsteczek siRNA z grupą cholesterolową lub pakowanie siRNA w cząsteczki liposomalne. Dostarczanie ogólnoustrojowe przy użyciu tych metod jest skuteczne w przypadku dostarczania do wątroby i jelita czczego, ale może nie być odpowiednie w przypadku dostarczania do innych narządów. W badaniu proof-of-concept zastosowano siRNA skierowane przeciwko apolipoproteinie B (APOB) w celu modyfikacji metabolizmu cholesterolu. Grupa hydroksylowa 3′ na nici pasażerskiej siRNA została chemicznie połączona z grupą cholesterolową, a te sprzężone siRNA skutecznie zmniejszyły ekspresję genu o >50% w wątrobie i 70% w jelicie czczym (26).
Inne podejście do ogólnoustrojowego dostarczania obejmuje wykorzystanie wyspecjalizowanych dwuwarstw lipidowych zwanych stabilnymi cząsteczkami kwasów nukleinowych-lipidów (SNALPs), które zawierają chemicznie zmodyfikowane siRNA (27). Kationowe i neutralne lipidy składają się na tę warstwę, wraz z zewnętrzną hydrofilową powłoką glikolu polietylenowego (PEG). W jednym z badań małpom podawano pojedynczą dawkę SNALPs zawierających siRNA, które obniżały poziom cholesterolu przez 11 dni lub dłużej, przy <10% ekspresji APOB pozostającej w wątrobie tego modelu naczelnych (28). Nie zaobserwowano żadnych zauważalnych toksyczności, co sugeruje potencjalną użyteczność tej metody w dostarczaniu ogólnoustrojowym.
Dla skuteczności in vivo cząsteczek siRNA, dawka dostarczonych siRNA jest praktycznym czynnikiem, a selektywne dostarczanie siRNA do określonych tkanek potencjalnie obniżyłoby wymaganą skuteczną dawkę. Celowanie w receptory powierzchniowe komórek jest korzystnym podejściem, ponieważ obniżyłoby dawkę siRNA i potencjalnie pozwoliłoby uniknąć efektów off-target wynikających z dostarczania siRNA do nieistotnych tkanek. Sprzężenie siRNA z aptamerami lub fragmentami przeciwciał, lub zastosowanie nanocząsteczek pokrytych ligandami specyficznymi dla receptorów, umożliwia specyficzne dostarczanie ładunku siRNA do docelowych komórek i tkanek. W celu ukierunkowania na komórki zakażone wirusem HIV, siRNA zostały sprzężone z fragmentami przeciwciał o ciężkim łańcuchu (Fabs), które rozpoznają glikoproteinę otoczki wirusa HIV gp120. Dodatnio naładowana protamina została sprzężona z cząsteczkami Fab, a ujemnie naładowane siRNA oddziaływały elektrostatycznie z protaminą, tworząc kompleks Fab-siRNA. To podejście oparte na przeciwciałach wykazało >70% spadek stężenia białka antygenu grupowego p24 (Gag) podczas celowania w hodowlane limfocyty T zakażone HIV-1 (29). Inne podejście do ukierunkowania wykorzystało peptyd z wirusa wścieklizny, który specyficznie wiąże się z receptorem acetylocholinowym. Kiedy ten peptyd został sprzężony z peptydem poliagininowym, który wiąże siRNA, udało się dostarczyć siRNA do centralnego układu nerwowego, co spowodowało zahamowanie śmiertelnej infekcji wirusowej zapalenia mózgu (30).
Aptamery, które są strukturalnymi ligandami RNA, mogą być zaprojektowane tak, aby wiązały się specyficznie z receptorami na powierzchni komórek i były kowalencyjnie połączone z siRNA w celu specyficznego dostarczenia in vivo. W jednej z metod wykorzystano aptamery, które wiążą się z antygenem błonowym specyficznym dla prostaty (PSMA), znajdującym się na powierzchni komórek raka prostaty. Po sprzężeniu z siRNA, te hybrydy aptamer-siRNA skutecznie zmniejszały wzrost guza u myszy (31). W podobnym podejściu, wykorzystującym zarówno biotynylowane siRNA, jak i aptamery związane z białkiem wiążącym biotynę – streptawidyną, wykorzystano 27-merowe siRNA do skutecznego wyciszania genów (32).
Pokrywanie nanocząsteczek ligandami specyficznymi dla danego typu komórek jest kolejnym skutecznym podejściem do systemowego dostarczania cząsteczek indukujących RNA. W ważnym badaniu proof-of-concept, guzy mięsaka Ewinga były celowane in vivo za pomocą nanocząstek pokrytych ligandem transferyny (33). Nanocząstki te zostały skonstruowane przy użyciu polikationów zawierających cyklodekstrynę (CDP), specjalnie zaprojektowanych do wbudowania ujemnie naładowanych cząsteczek siRNA. W celu zwiększenia stabilności i zapobieżenia agregacji, polimery PEG zostały dołączone do zewnętrznej powierzchni za pomocą terminalnych grup adamantanowych. Ligandy transferyny zostały następnie kowalencyjnie połączone z łańcuchami adamantan-PEG, a konstrukcja nanocząstek pozwoliła na samoczynne złożenie się w jednolite nanocząstki o rozmiarach ∼50 nanometrów. Wprowadzone do nanocząstek siRNA celowało w produkt fuzji genów Ews-Fli1 (Ewing sarcoma breakpoint region 1-flightless 1 homolog) i wykazało, że hamowało tworzenie się guzów u myszy (33).
Uwagi końcowe
Podsumowując, postęp od początkowego odkrycia RNAi do jego zastosowań klinicznych był zdumiewający. Zrozumienie fundamentalnej biologii RNAi doprowadziło do jego szerokiego zastosowania w badaniach podstawowych, a następnie w leczeniu chorób. W ciągu najbliższych kilku lat powinniśmy spodziewać się odkrycia kolejnych mechanizmów regulacji ekspresji genów za pomocą RNAi, a także leków opartych na RNAi dopuszczonych do stosowania w leczeniu chorób. Ponadto, RNAi okazało się być potężnym narzędziem do badania funkcji genów i otworzyło nowe obszary badań podstawowych. W najbliższej przyszłości powinniśmy zobaczyć ciągły rozwój w naszym zrozumieniu i zastosowaniu tego niezwykłego mechanizmu komórkowego do posttranskrypcyjnej regulacji ekspresji genów.
Podziękowania
Ta praca była wspierana przez National Institutes of Health National Institute of Allergy and Infectious Diseases i NIH Heart Lung and Blood Institute dla J.J.R. D.H.K. jest wspierany przez City of Hope pre-doctoral fellowship.
Oświadczenie o interesach konkurencyjnych
Autorzy deklarują brak interesów konkurencyjnych.
- 1. Almeida, R. i R.C. Allshire. 2005. Wyciszanie RNA i regulacja genomu. Trends Cell Biol. 15:251-258.Crossref, Medline, CAS, Google Scholar
- 2. Zhang, H., F.A. Kolb, L. Jaśkiewicz, E. Westhof, and W. Filipowicz. 2004. Single processing center models for human Dicer and bacterial RNase III. Cell 118:57-68.Crossref, Medline, CAS, Google Scholar
- 3. Lee, Y., I. Hur, S.Y. Park, Y.K. Kim, M.R. Suh, and V.N. Kim. 2006. The role of PACT in the RNA silencing pathway. EMBO J. 25:522-532.Crossref, Medline, CAS, Google Scholar
- 4. Meister, G., M. Landthaler, A. Patkaniowska, Y. Dorsett, G. Teng, and T. Tuschl. 2004. Human Argonaute2 mediates RNA cleavage targeted by miRNAs and siRNAs. Mol. Cell 15:185-197.Crossref, Medline, CAS, Google Scholar
- 5. Liu, J., M.A. Carmell, F.V. Rivas, C.G. Marsden, J.M. Thomson, J.J. Song, S.M. Hammond, L. Joshua-Tor, et al. 2004. Argonaute2 is the catalytic engine of mammalian RNAi. Science 305:1437-41.Crossref, Medline, CAS, Google Scholar
- 6. Tang, G. 2005. siRNA and miRNA: an insight into RISCs. Trends Biochem. Sci. 30:106-114.Crossref, Medline, CAS, Google Scholar
- 7. Schwarz, D.S., G. Hutvagner, T. Du, Z. Xu, N. Aronin, and P.D. Zamore. 2003. Asymmetry in the assembly of the RNAi enzyme complex. Cell 115:199-208.Crossref, Medline, CAS, Google Scholar
- 8. Khvorova, A., A. Reynolds, and S.D. Jayasena. 2003. Functional siRNAs and miRNAs exhibit strand bias. Cell 115:209-216.Crossref, Medline, CAS, Google Scholar
- 9. Bartel, D.P. 2004. MikroRNA: genomika, biogeneza, mechanizm i funkcja. Cell 116:281-297.Crossref, Medline, CAS, Google Scholar
- 10. Bartel, D.P. i C.Z. Chen. 2004. Micromanagers of gene expression: the potentially widespread influence of metazoan microRNAs. Nat. Rev. Genet. 5:396-400.Crossref, Medline, CAS, Google Scholar
- 11. Bagga, S., J. Bracht, S. Hunter, K. Massirer, J. Holtz, R. Eachus, and A.E. Pasquinelli. 2005. Regulation by let-7 and lin-4 miRNAs results in target mRNA degradation. Cell 122:553-563.Crossref, Medline, CAS, Google Scholar
- 12. Leuschner, P.J., S.L. Ameres, S. Kueng, and J. Martinez. 2006. Cleavage of the siRNA passenger strand during RISC assembly in human cells. EMBO Rep. 7:314-320.Crossref, Medline, CAS, Google Scholar
- 13. Gregory, R.I., T.P. Chendrimada, N. Cooch, and R. Shiekhattar. 2005. Human RISC couples microRNA biogenesis and posttranscriptional gene silencing. Cell 123:631-640.Crossref, Medline, CAS, Google Scholar
- 14. Matranga, C., Y. Tomari, C. Shin, D.P. Bartel, and P.D. Zamore. 2005. Passenger-strand cleavage facilitates assembly of siRNA into Ago2-containing RNAi enzyme complexes. Cell 123:607-620.Crossref, Medline, CAS, Google Scholar
- 15. Lee, Y., C. Ahn, J. Han, H. Choi, J. Kim, J. Yim, J. Lee, P. Provost, et al. 2003. The nuclear RNase III Drosha initiates microRNA processing. Nature 425:415-419.Crossref, Medline, CAS, Google Scholar
- 16. Han, J., Y. Lee, K.H. Yeom, Y.K. Kim, H. Jin, and V.N. Kim. 2004. The Drosha-DGCR8 complex in primary microRNA processing. Genes Dev. 18:3016-3027.Crossref, Medline, CAS, Google Scholar
- 17. Hannon, G.J. i J.J. Rossi. 2004. Odblokowanie potencjału ludzkiego genomu za pomocą interferencji RNA. Nature 431:371-378.Crossref, Medline, CAS, Google Scholar
- 18. Scherer, L.J. i J.J. Rossi. 2003. Approaches for the sequence-specific knockdown of mRNA. Nat. Biotechnol. 21:1457-1465.Crossref, Medline, CAS, Google Scholar
- 19. Fattal, E. i A. Bochot. 2006. Ocular delivery of nucleic acids: antisense oligonucleotides, aptamers and siRNA. Adv. Drug Deliv. Rev. 58:1203-1223.Crossref, Medline, CAS, Google Scholar
- 20. Tolentino, M. 2006. Technologia interferencyjnego RNA w leczeniu CNV. Ophthalmol. Clin. North Am. 19:393-399 vi-vii.Medline, Google Scholar
- 21. Leonard, J.N. i D.V. Schaffer. 2006. Terapia przeciwwirusowa RNAi: pojawiające się podejścia do uderzania w ruchomy cel. Gene Ther. 13:532-540.Crossref, Medline, CAS, Google Scholar
- 22. Svoboda, P. 2007. Off-targeting i inne niespecyficzne efekty eksperymentów RNAi w komórkach ssaków. Curr. Opin. Mol. Ther. 9:248-257.Medline, CAS, Google Scholar
- 23. Czauderna, F., M. Fechtner, S. Dames, H. Aygun, A. Klippel, G.J. Pronk, K. Giese, and J. Kaufmann. 2003. Structural variations and stabilising modifications of synthetic siRNAs in mammalian cells. Nucleic Acids Res. 31:2705-2716.Crossref, Medline, CAS, Google Scholar
- 24. Kim, D.H., M.A. Behlke, S.D. Rose, M.S. Chang, S. Choi, and J.J. Rossi. 2005. Synthetic dsRNA Dicer substrates enhance RNAi potency and efficacy. Nat. Biotechnol. 23:222-226.Crossref, Medline, CAS, Google Scholar
- 25. Amarzguioui, M., P. Lundberg, E. Cantin, J. Hagstrom, M.A. Behlke, and J.J. Rossi. 2006. Rational design and in vitro and in vivo delivery of Dicer substrate siRNA. Nat. Protocols 1:508-517.Crossref, Medline, CAS, Google Scholar
- 26. Soutschek, J., A. Akinc, B. Bramlage, K. Charisse, R. Constien, M. Donoghue, S. Elbashir, A. Geick, et al. 2004. Therapeutic silencing of an endogenous gene by systemic administration of modified siRNAs. Nature 432:173-178.Crossref, Medline, CAS, Google Scholar
- 27. Morrissey, D.V., J.A. Lockridge, L. Shaw, K. Blanchard, K. Jensen, W. Breen, K. Hartsough, L. Machemer, et al. 2005. Potent and persistent in vivo anti-HBV activity of chemically modified siRNAs. Nat. Biotechnol. 23:1002-1007.Crossref, Medline, CAS, Google Scholar
- 28. Zimmermann, T.S., A.C. Lee, A. Akinc, B. Bramlage, D. Bumcrot, M.N. Fedoruk, J. Harborth, J.A. Heyes, et al. 2006. RNAi-mediated gene silencing in non-human primates. Nature 441:111-114.Crossref, Medline, CAS, Google Scholar
- 29. Song, E., P. Zhu, S.K. Lee, D. Chowdhury, S. Kussman, D.M. Dykxhoorn, Y. Feng, D. Palliser, et al. 2005. Antibody mediated in vivo delivery of small interfering RNAs via cell-surface receptors. Nat. Biotechnol. 23:709-717.Crossref, Medline, CAS, Google Scholar
- 30. Kumar, P., H. Wu, J.L. McBride, K.E. Jung, M.H. Kim, B.L. Davidson, S.K. Lee, P. Shankar, et al. 2007. Transvascular delivery of small interfering RNA to the central nervous system. Nature 448:39-43.Crossref, Medline, CAS, Google Scholar
- 31. McNamara, J.O., II, E.R. Andrechek, Y. Wang, K.D. Viles, R.E. Rempel, E. Gilboa, B.A. Sullenger, and P.H. Giangrande. 2006. Cell-specific delivery of siRNAs with aptamer-siRNA chimeras. Nat. Biotechnol. 24:1005-1015.Crossref, Medline, CAS, Google Scholar
- 32. Chu, T.C., K.Y. Twu, A.D. Ellington, and M. Levy. 2006. Aptamer mediated siRNA delivery. Nucleic Acids Res. 34:e73.Crossref, Medline, Google Scholar
- 33. Hu-Lieskovan, S., J.D. Heidel, D.W. Bartlett, M.E. Davis, and T.J. Triche. 2005. Sequence-specific knockdown of EWS-FLI1 by targeted, nonviral delivery of small interfering RNA inhibits tumor growth in a murine model of metastatic Ewing’s s sarcoma. Cancer Res. 65:8984-8992.Crossref, Medline, CAS, Google Scholar